估计阅读时长: 2 分钟Docker镜像信息 GCModeller以R#语言的软件包的形式提供给客户使用,相应的R#语言的分析环境以Docker镜像的形式进行打包盒发布,Docker的基础镜像为ubuntu 22.04。 dotnet环境:.NET 6 R#语言安装位置:/usr/local/bin R#程序包安装列表: 索引 包名称 Github 1 GCModeller https://github.com/SMRUCC/GCModeller 2 REnv https://github.com/rsharp-lang/R-sharp […]
估计阅读时长: 7 分钟一般而言,进行全基因组的转录表达调控网络的建立,我们需要基于两个数据结果来完成: 目标基因的转录调控位点信息(Motif搜索结果,构成网络之中的节点) 转录调控位点相应的转录调控因子(Motif位点相关的转录调控因子,构成网络之中的边连接) Order by Date Name Attachments Xor • 271 kB • 246 click 2022年6月11日An […]
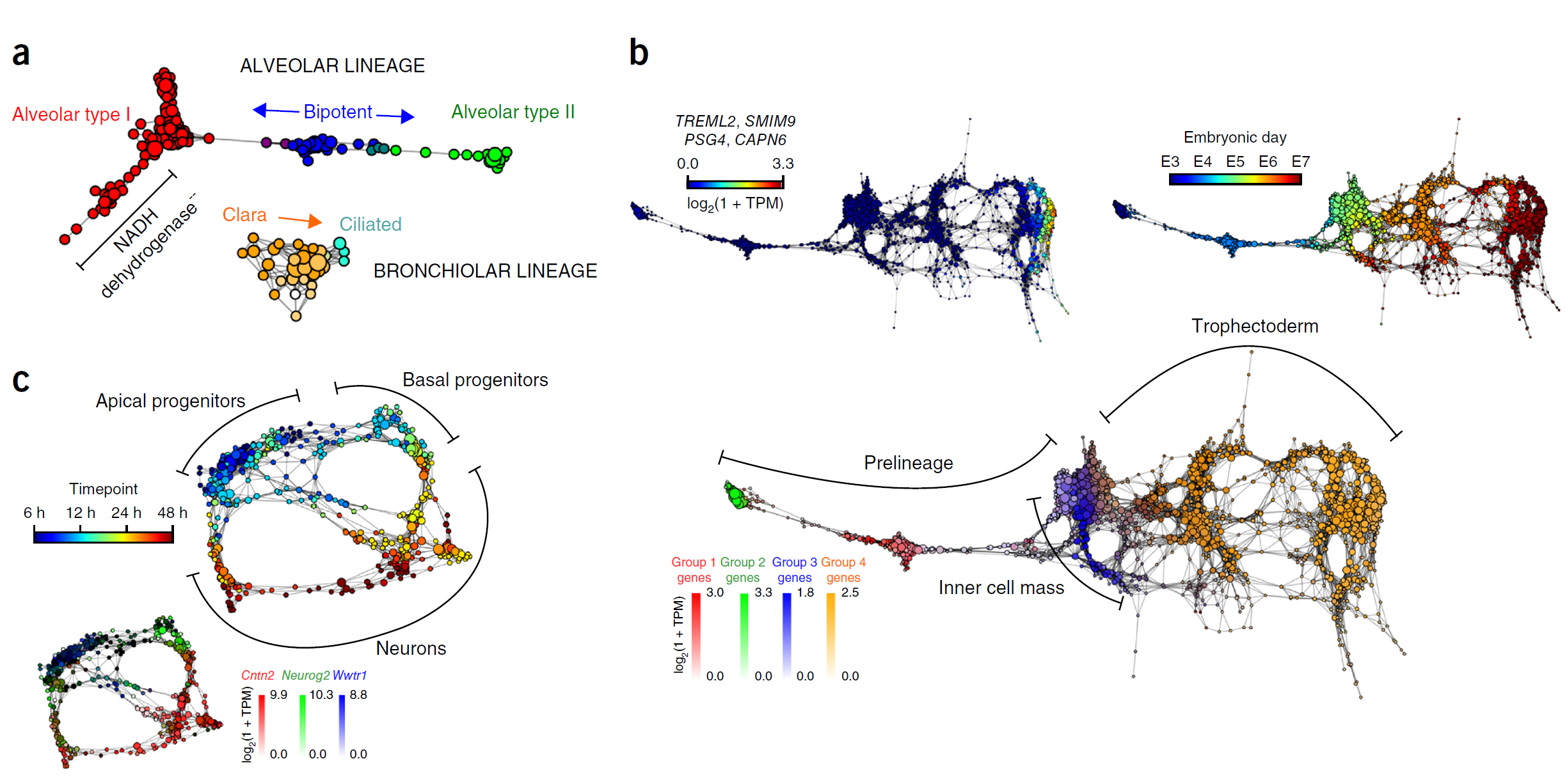
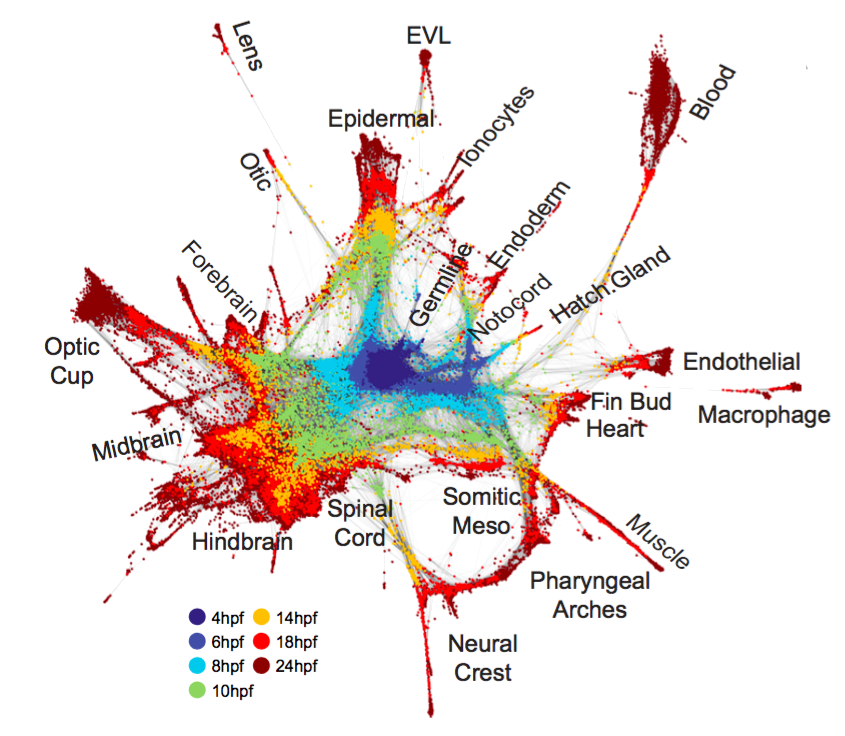
估计阅读时长: 14 分钟单细胞分析方法学习文献打卡记录: 【单细胞组学】PhenoGraph单细胞分型 【单细胞分析方法】VeTra:基于RNA速度的轨迹推断工具 【单细胞分析方法】单细胞图嵌入 Order by Date Name Attachments Cellular populations during motor neuron differentiation • […]
估计阅读时长: 7 分钟Assembles a manifold that is defined through a series of overlapping, locally-defined PCA subspaces. Non-mutual k-nearest-neighborhoods […]
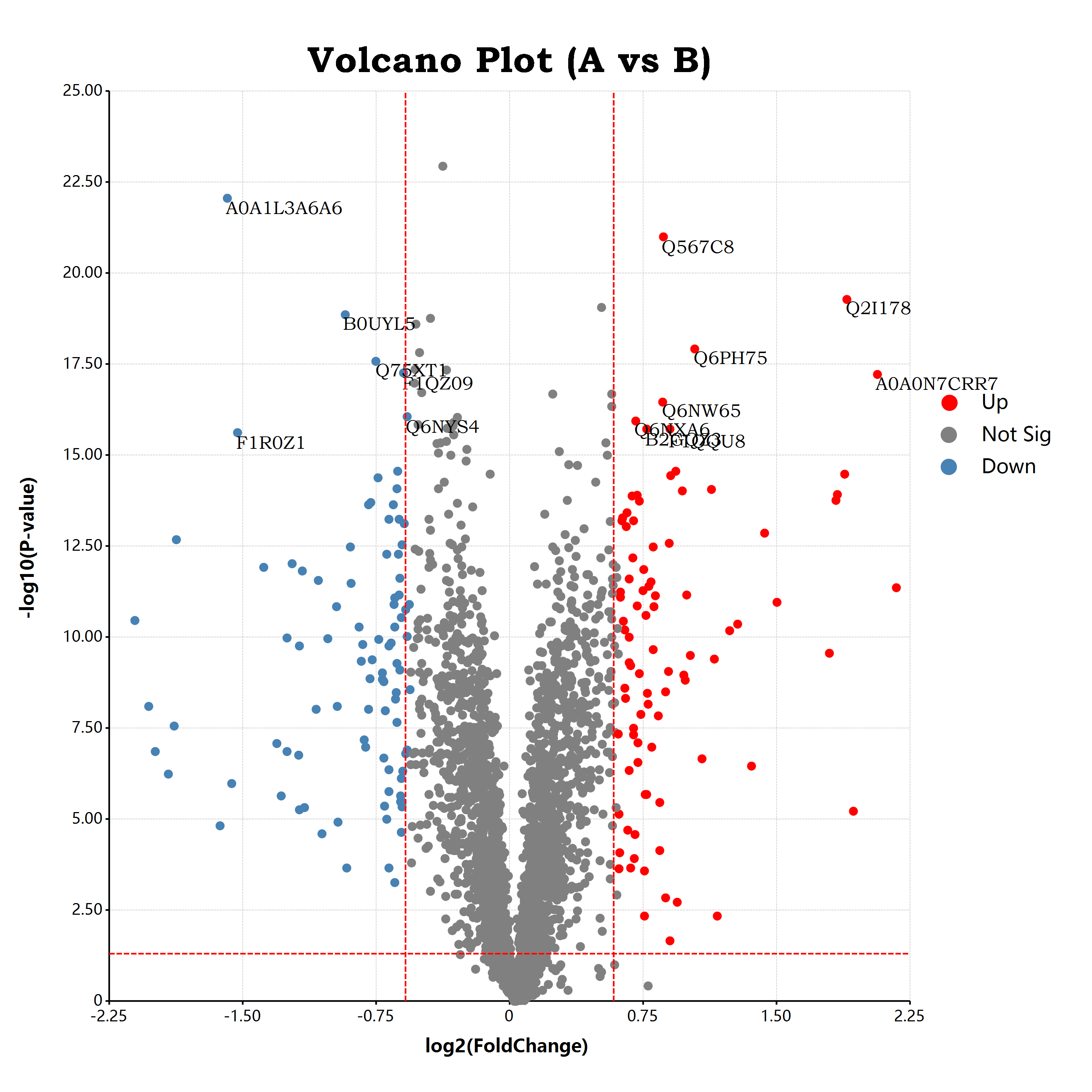
估计阅读时长: 17 分钟https://github.com/rsharp-lang/ggplot 接上一篇博客文章中谈到,我们已经通过R#语言之中的ggplot程序包绘制出了一个可以使用的火山图。在这里,我们将会通过在火山图上添加更多的可视化元素来为大家介绍R#语言之中的ggplot程序包的进阶使用方式。 Order by Date Name Attachments volcano • 651 kB • 296 click 2021年10月9日volcano • […]
估计阅读时长: 11 分钟https://github.com/rsharp-lang/ggplot 在生物信息学中的组学数据分析领域内,有一个非常常见的数据可视化图表:应用于可视化两两组别比对结果的火山图。在火山图之中,X坐标轴一般是log2FC,纵坐标Y轴,则一般是t检验的pvalue的-log10转换之后的值。由于fold change有大于1的值,A/B大于1,表示A的表达量高于B的表达量,反之小于一表示A的表达量低于B的表达量。这样子fold change经过log2转换之后,就会出现负数,散点一般呈轴对称分布在X=0的位置周围。这样子绘制出来的散点图就有点类似于火山喷发的样子了。 Order by Date Name Attachments a679af1eb9ffbfbad48c18d563ea51f3 • 45 kB • 320 click […]
估计阅读时长: 15 分钟https://gcmodeller.org 在这篇博客文章之中,我主要是来详细介绍一下是如何从头开始实现Phenograph单细胞分型算法的。在之前的一篇博客文章《【单细胞组学】PhenoGraph单细胞分型》之中,我们介绍了Phenograph算法的简单原理,以及一个在R语言之中所实现的Phenograph算法的程序包Rphenograph。在这里我主要是详细介绍在GCModeller软件之中所实现的VisualBasic语言版本的Phenograph单细胞分型算法。 Attachments Rphenograph • 236 kB • 237 click 2021年9月20日
估计阅读时长: 8 分钟https://gcmodeller.org/ KEGG is a database resource for understanding high-level functions and utilities of the biological system, […]
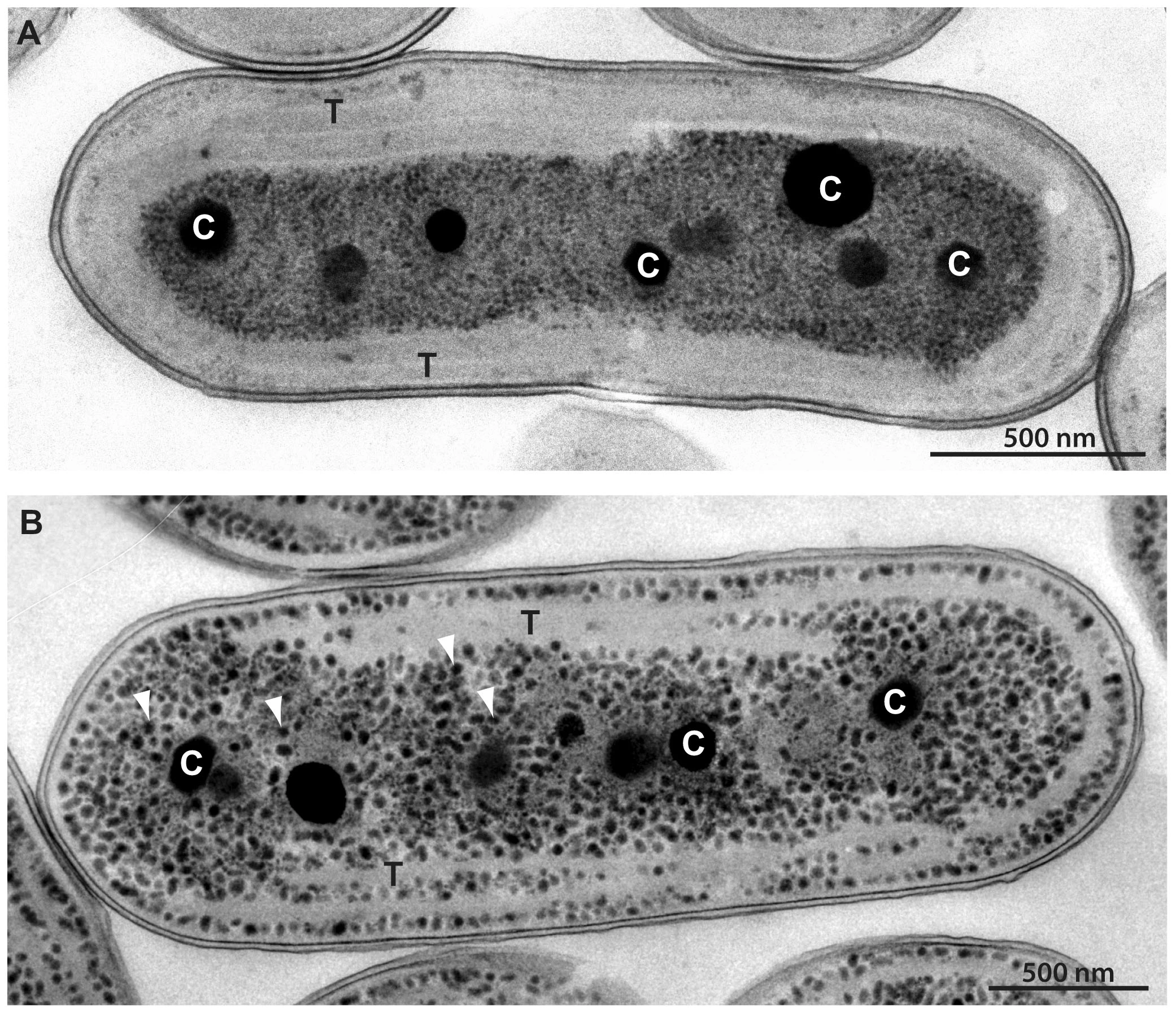
估计阅读时长: 10 分钟https://gcmodeller.org/ 流平衡分析(flux balance analysis)是一种可以用来构建和模拟分析基因组级别的代谢网络的数学方法。流平衡分析是系统生物学(system biology)的一个重要的分析手段。不同于以湿实验的代谢通量分析(metabolic flux analysis, MFA),FBA是用数学方法对代谢网络里的代谢流进行拟合分析。 Order by Date Name Attachments Electron micrographs of […]




Hello There. I found your website through the usage of msn. This is a very smartly written article. I will…
[…] […]
[…] […]
[…] 《为大语言模型运行添加工具调用》 […]
[…] 《从头创建一个DeepSeek客户端》 […]