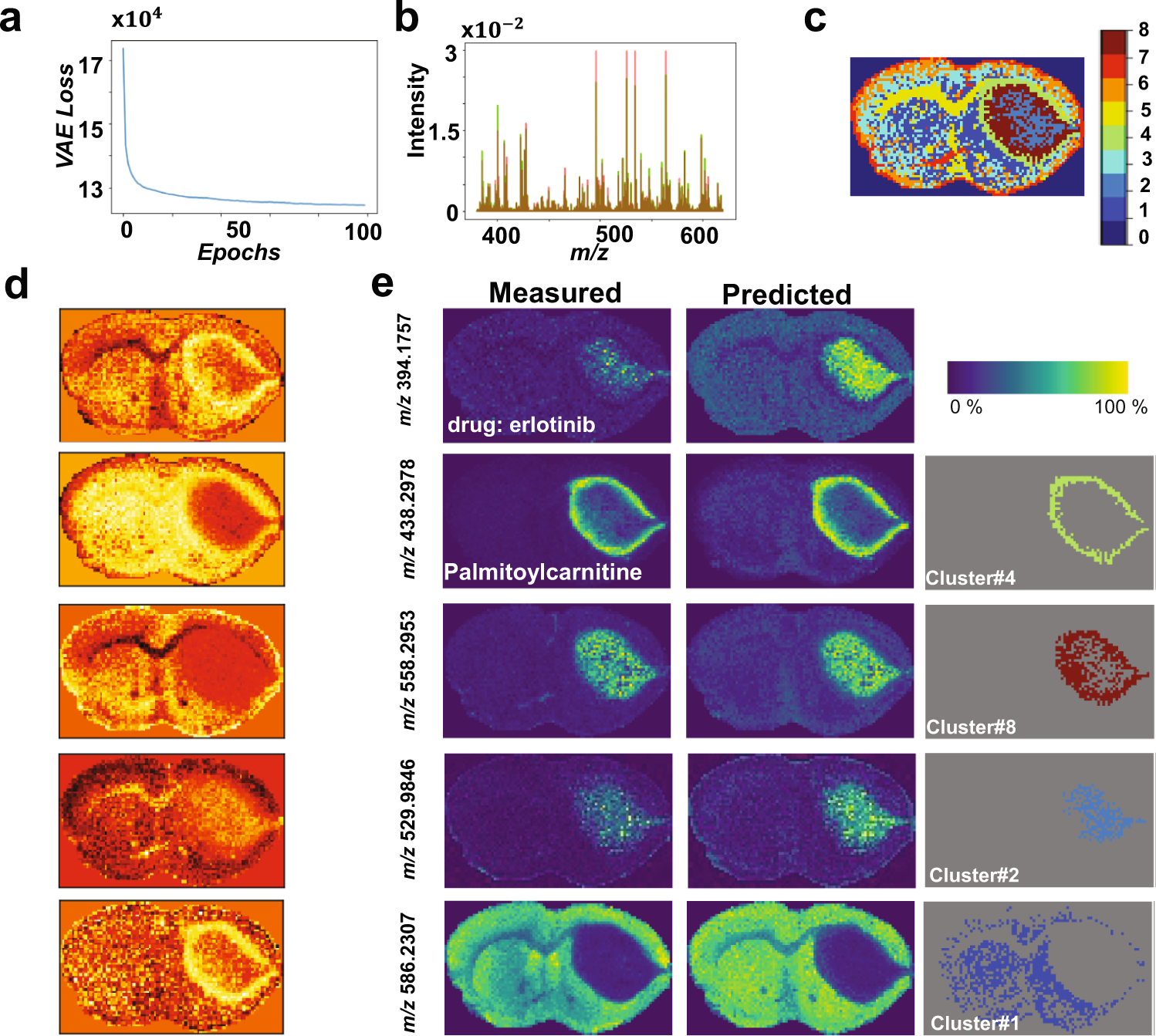
估计阅读时长: 4 分钟基于UMAP工具进行简单的自动化组织分区操作 在这里我们假设已经可以正常的将空间代谢数据导入至MZKit工作站软件之中。假若需要借助于MZKit工作站软件进行切片组织样本的自动化分区操作,相关的功能可以在【MSI Analysis】菜单栏中寻找到。在这里我们打开【Show Map Layer】按钮,选择【UMAP and clustering】功能。 基于降维的组织自动化分区原理 因为降维操作一般是一种特征提取操作,所以经过降维之后,在高维度空间上无法显现的特征,在低维度会呈现出来。在高维度空间散落的相近的数据点,在经过特征提取之后,低维度上会产生相似的特征信息,相互聚集在一簇。这样子我们就可以在低维度空间上通过一些聚类算法讲这些特征进行聚类,最后将聚类特征结果标记到各个散点上的对应的原始成像空间上,我们就可以看见组织分区的结果了。 Abdelmoula, W.M., Lopez, B.GC., Randall, E.C. et […]
估计阅读时长: 6 分钟大家好呀,今天的这篇文章主要是为了回答在B站上的一位小伙伴的请求 Order by Date Name Attachments render-parameters • 18 kB • 207 click 2023年10月15日view-umap • 427 […]
估计阅读时长: 5 分钟在BILIBILI上观看视频:《【BioNovoGene Mzkit教程】代谢组学原始数据处理基础》 最近我在B站的视频页面下发现了这样的一条评论,面对质谱数据分析领域内的初学者的求教,其实自己也是非常的诚惶诚恐的。因为在视频中所使用的脚本语言是自己开发的一门新语言,所以可能给一些初学者造成了一部分的困扰哈哈😅😄😅😅。首先先对这个粉丝说一声抱歉哈。 针对上述的提问,我的回答大概是有以下的几点: Order by Date Name Attachments question_20230223 • 17 kB • 176 click […]
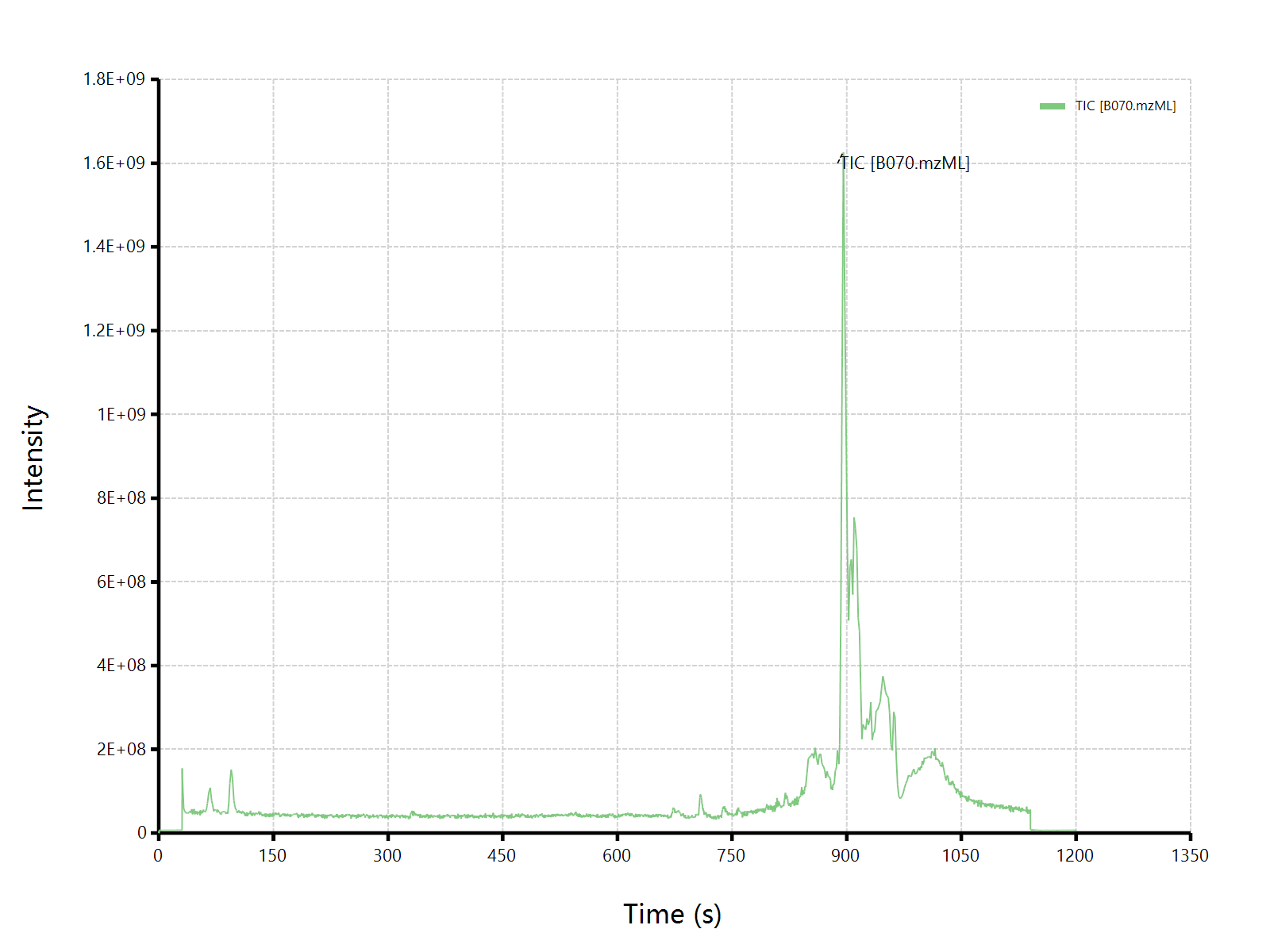
估计阅读时长: 4 分钟在代谢组学领域内,LCMS原始数据分析一般分为非靶向全扫原始数据,以及仅针对某些离子进行扫描的MRM靶向质谱数据。虽然二者都是基于LCMS方法进行实验,但是MRM靶向数据由于在事先已经通过实验确定,得到了Q1和Q3离子对信息,所以可以仅针对某一些特定代谢物进行检测。因为MRM数据是针对于某些代谢物检测的靶向数据,所以其XIC谱图在没有同分异构体存在的情况下,一般是很纯净的目标化合物的检测结果数据。所以在原始数据分离,定量计算方面都要比非靶向全扫结果数据要容易很多。 Order by Date Name Attachments xcms-logo-white • 183 kB • 255 click 2022年7月1日lcmspreproc_slides_1.2 • 136 […]
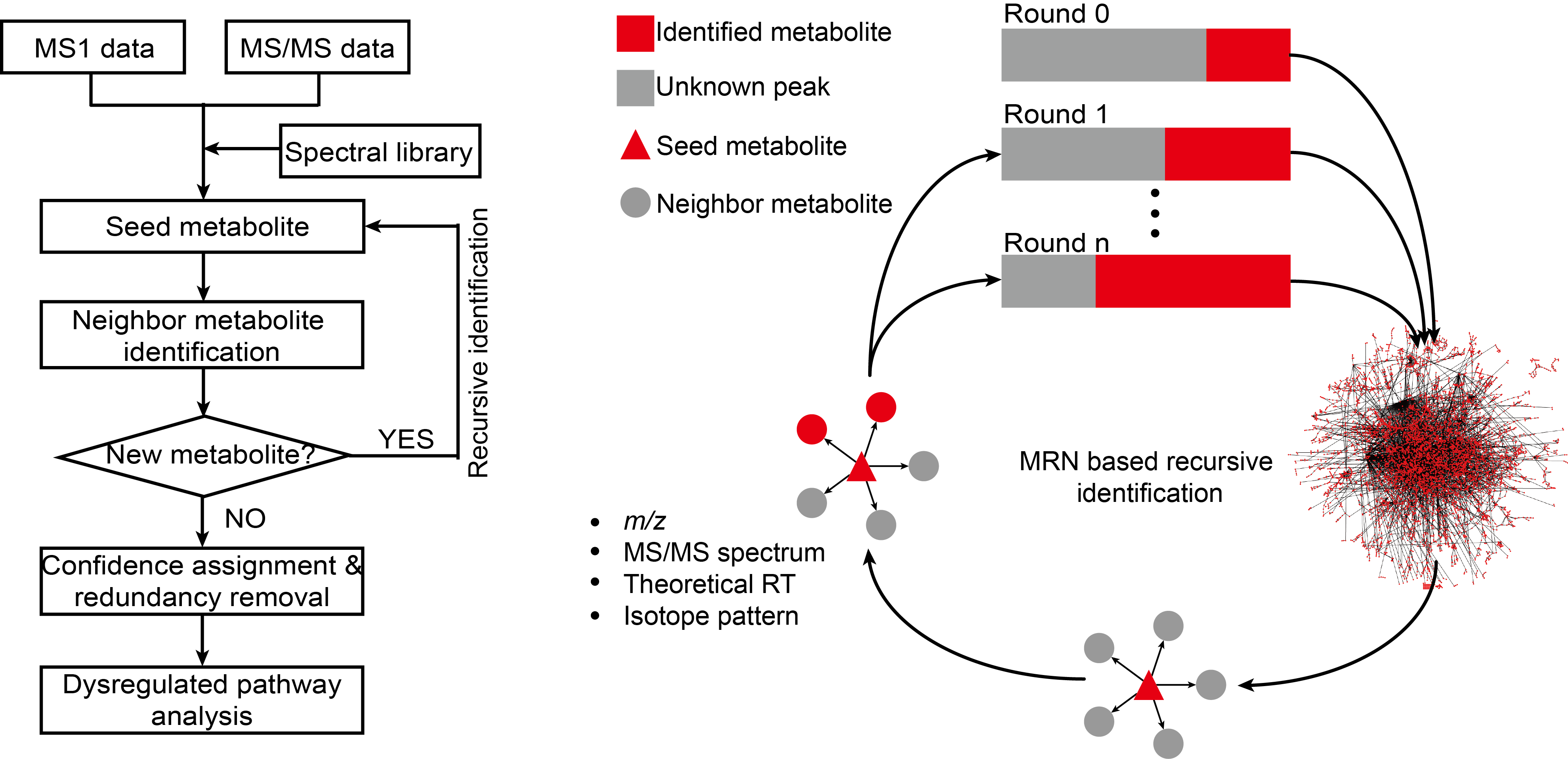
估计阅读时长: 5 分钟目前我们根据质谱数据进行代谢物ROI注释分析,很大一部分的工作是建立在已经可以被纯化的化合物的纯标准品所建立的标准品库数据的比对操作之上的。但是依赖于质谱参考谱图数据库所完成的代谢物注释分析,也仅能够得到很小的一部分结果,因为能够纯化或者合成的化合物在整个自然界中目前只占比较小的一部分。并且购买标准品也会需要耗费大量的实验室资金预算。 Order by Date Name Attachments The-Periodic-Table • 2 MB • 330 click 2022年3月20日Leucine[M+H]+ • 33 […]
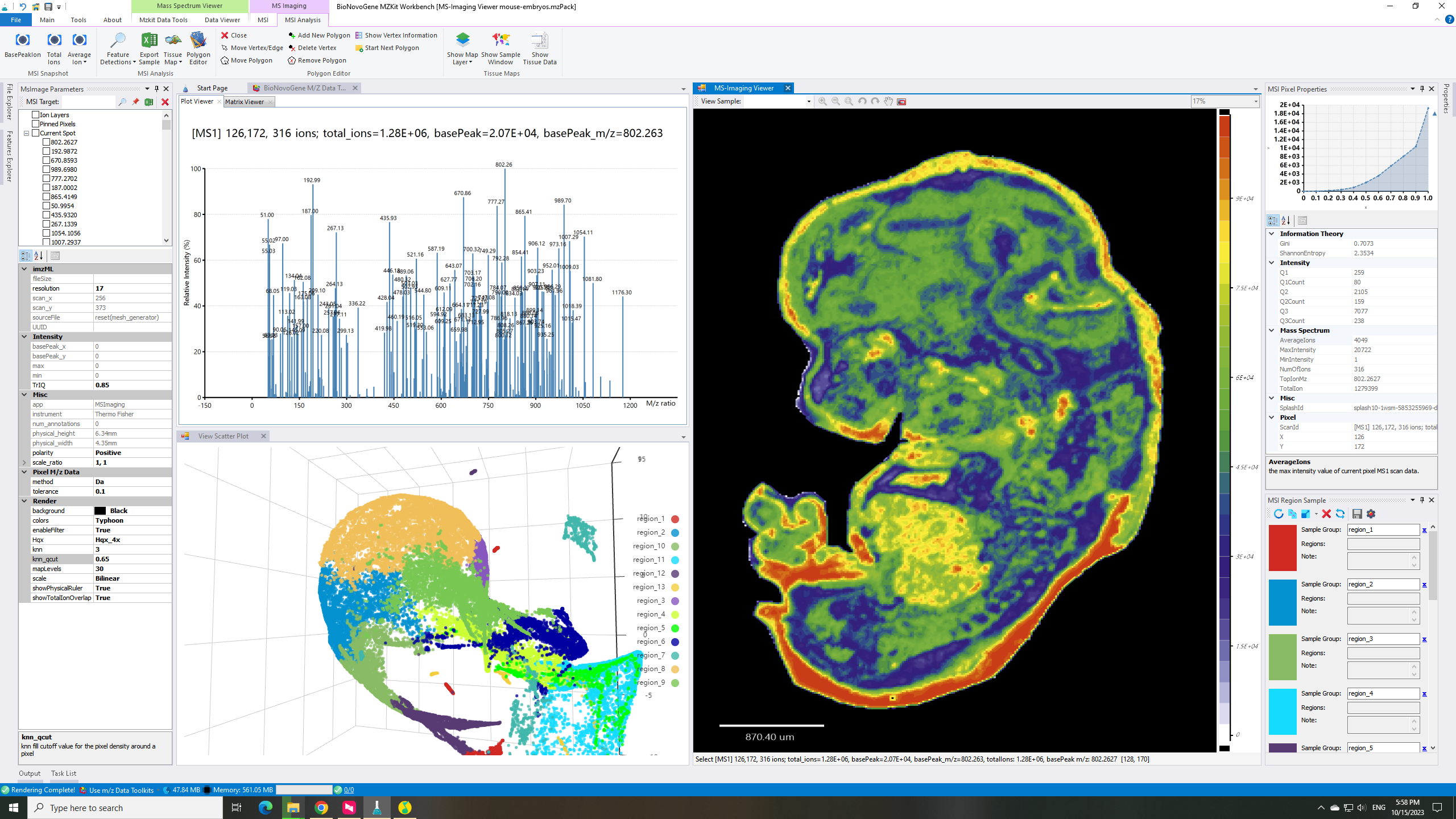
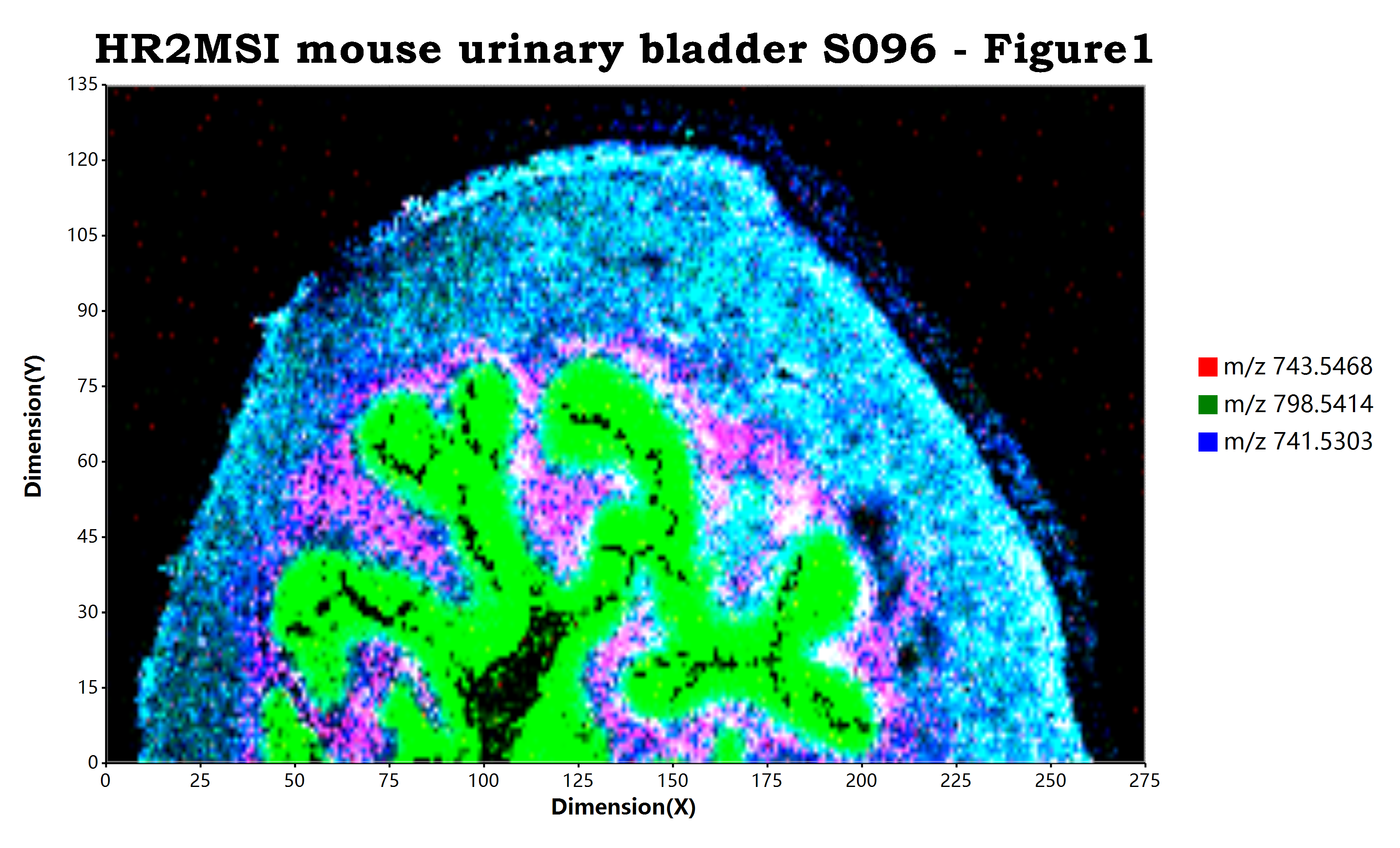
估计阅读时长: 10 分钟https://github.com/xieguigang/ms-imaging Order by Date Name Attachments HR2MSI_mouse_urinary_bladder_S096_RGB • 7 MB • 277 click 2021年11月13日peerj-cs-07-585 • 16 […]
估计阅读时长: 11 分钟https://github.com/xieguigang/ms-imaging Mass spectrometry imaging ( MSI) is a technique used in mass spectrometry to visualize the […]
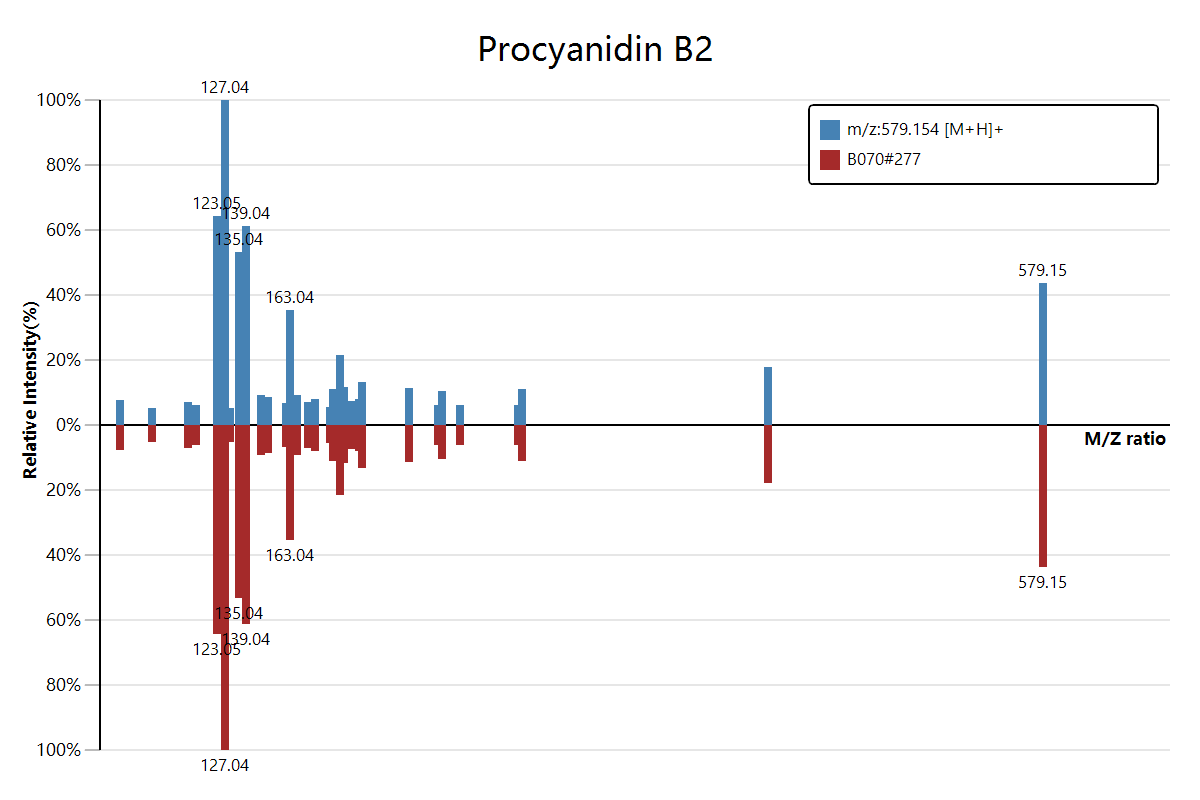
估计阅读时长: 2 分钟https://mzkit.org/ 代谢组文章整个坎坷的发表经历中,大家可能都会遇到的一个老大难问题就是我们有时候会需要从原始数据中得到物质注释结果的二级质谱图数据。对于熟悉xcms程序包的同学,获取二级质谱图可能会比较容易:无非就是加载原始数据,然后按照m/z和rt找到对应的二级scan就好了。但是,这种方法会需要编写脚本来完成。 Order by Date Name Attachments download • 33 kB • 286 click 2021年8月4日[278][MS_MS] FTMS […]
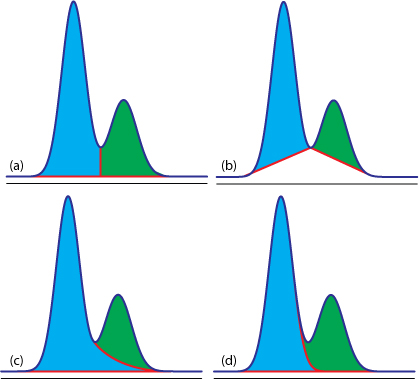
估计阅读时长: 8 分钟https://github.com/xieguigang/sciBASIC/tree/master/Data_science/Mathematica/SignalProcessing 进行峰识别是在代谢组学原始数据分析之中进行定量分析的很重要的一环。在代谢组学之中,定量分析分为靶向定量,以及非靶向定量计算这两大部分。 Order by Date Name Attachments Figure12.36 • 50 kB • 300 click 2021年7月10日view_signal • […]




Hello There. I found your website through the usage of msn. This is a very smartly written article. I will…
[…] […]
[…] […]
[…] 《为大语言模型运行添加工具调用》 […]
[…] 《从头创建一个DeepSeek客户端》 […]