估计阅读时长: 2 分钟https://github.com/dotvanilla/vanilla 在Vanilla编译器项目之中,会需要一个程序模块将VisualBasic代码进行解析为语法树。然后我们基于此语法树就可以将VisualBasic项目转换为WAST源代码,从而实现编译为WebAssembly程序了。在这个步骤之中,我们可以通过一个微软官方的Roslyn编译器平台来实现。 Order by Date Name Attachments Roslyn-nuget • 107 kB • 278 click 2021年7月24日what-is-visual-studio • […]
估计阅读时长: < 1 分钟https://github.com/xieguigang/codegraph Attachments Microsoft.VisualBasic.Framework_v47_dotnet_8da45dcd8060cc9a.dll • 10 MB • 223 click 2021年8月29日
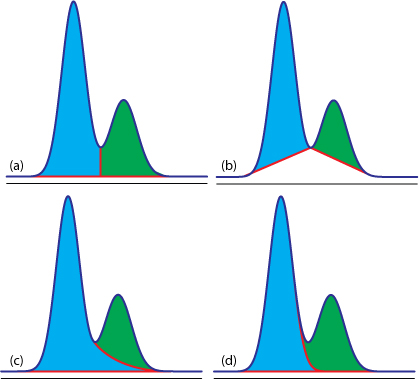
估计阅读时长: 8 分钟https://github.com/xieguigang/sciBASIC/tree/master/Data_science/Mathematica/SignalProcessing 进行峰识别是在代谢组学原始数据分析之中进行定量分析的很重要的一环。在代谢组学之中,定量分析分为靶向定量,以及非靶向定量计算这两大部分。 Order by Date Name Attachments Figure12.36 • 50 kB • 300 click 2021年7月10日view_signal • […]
估计阅读时长: 9 分钟https://github.com/dotvanilla/vanilla WebAssembly是一种运行在浏览器端的二进制程序集文件。和普通的应用程序开发一样,WebAssembly需要基于一定的源代码文本进行编译。这个编译所需要的源代码文本就是WAST文件。 Understanding WebAssembly text format(https://developer.mozilla.org/en-US/docs/WebAssembly/Understanding_the_text_format)
估计阅读时长: 4 分钟https://github.com/dotvanilla/vanilla vanilla编译器项目是我之前开发过的一个实验性质的项目。主要是为了解决在浏览器端的一些高性能计算的需求,例如数据加密和解密,基于WebGL的计算机图形项目,力学物理规律模拟,网络可视化布局计算等。 Order by Date Name Attachments 1_PcKt44c-UZBBTfNBaovxeQ • 49 kB • 229 click 2021年7月8日web-assembly-architecture-xenonstack-3-1 • […]
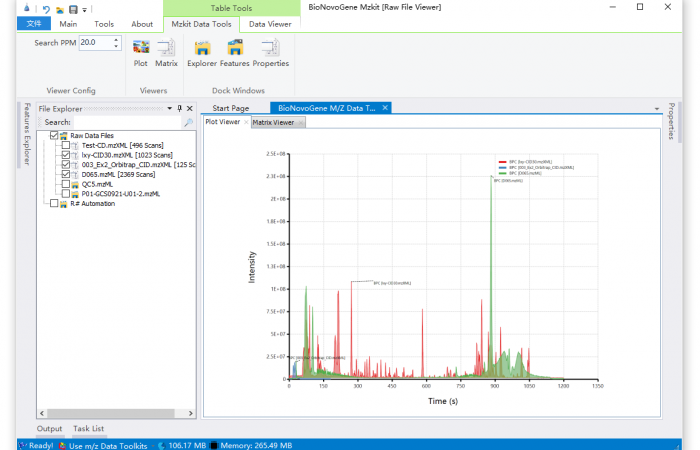
估计阅读时长: 3 分钟https://mzkit.org mzkit软件是我最近开发的一款开源的代谢组学领域内的原始数据文件查看工具。开发mzkit软件的初衷是为了更方便的查看很大的非靶向原始数据文件:因为在开发mzkit软件之前,在开发LCMS的代谢物注释脚本或者建立标准品库数据的时候,如果我想要查看或者导出文件中的一些质谱图碎片信息,会需要通过R环境之中的xcms程序包编程来完成。通过R脚本来查看原始数据文件,非常的不方便。所以就有了mzkit软件项目的诞生。 Order by Date Name Attachments BPC_overlay • 114 kB • 331 click 2021年7月1日LCMS_scanTree • […]




Hello There. I found your website through the usage of msn. This is a very smartly written article. I will…
[…] […]
[…] […]
[…] 《为大语言模型运行添加工具调用》 […]
[…] 《从头创建一个DeepSeek客户端》 […]