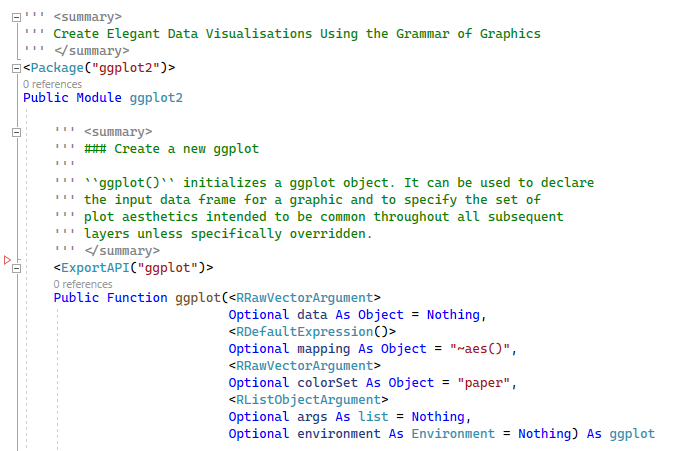
估计阅读时长: 9 分钟因为一种单一的编程语言并不会覆盖到所有的适用场景的原因,在一个软件工程项目之中,采用多种语言进行混合编程是一种很常见的协作方式。例如,脚本化的语言,其非常适合于进行最顶层的应用开发,就像胶水一样用于将各种组件进行粘贴,但是脚本化的语言自身因为是基于其他的语言所构建,所以执行效率一般不会太好。对于底层组件,我们一般就会需要使用静态编译类型的非托管语言创建用于高性能数据处理的模块。对于这种需求的底层模块,我们一般可以采用C/C++/Rust来编写。 Order by Date Name Attachments rust • 162 kB • 200 click 2023年3月25日dyn-load • 67 […]
估计阅读时长: 31 分钟Like the original R language it does, the R# system just provides a runtime to running […]
估计阅读时长: 9 分钟https://github.com/rsharp-lang/Rnb 之前使用Python脚本进行编写代码的时候,十分的羡慕Python脚本可以基于ipynb记事本进行文档化的编码。在之前R#脚本是缺少相关的代码库模块将可执行的R#脚本渲染成可视化文档。但是经过几天的开发工作时候,现在R#脚本编程已经具备有了文档化编程的基本框架了。 Order by Date Name Attachments 01510007-school-notebook • 32 kB • 250 click 2021年10月30日renderHtml_cli • […]
估计阅读时长: 6 分钟之前在阅读一个使用rust语言编写的contour tracing算法模块的源代码的时候,其中有一个向量的左旋以及右旋的操作。这个操作的具体的含义是和在算法中的轮廓边缘像素的读取方向有关:因为访问方向是一个二维平面的概念,但是在代码中我们只能够使用一个一维的数组的来存储这个二维的信息。所以在这段rust代码之中,作者很巧妙的使用了向量的左旋以及右旋操作来实现一维数组中对二维平面上的方位的访问操作。 Order by Date Name Attachments RotateVector • 30 kB • 251 click 2021年9月16日Full • […]
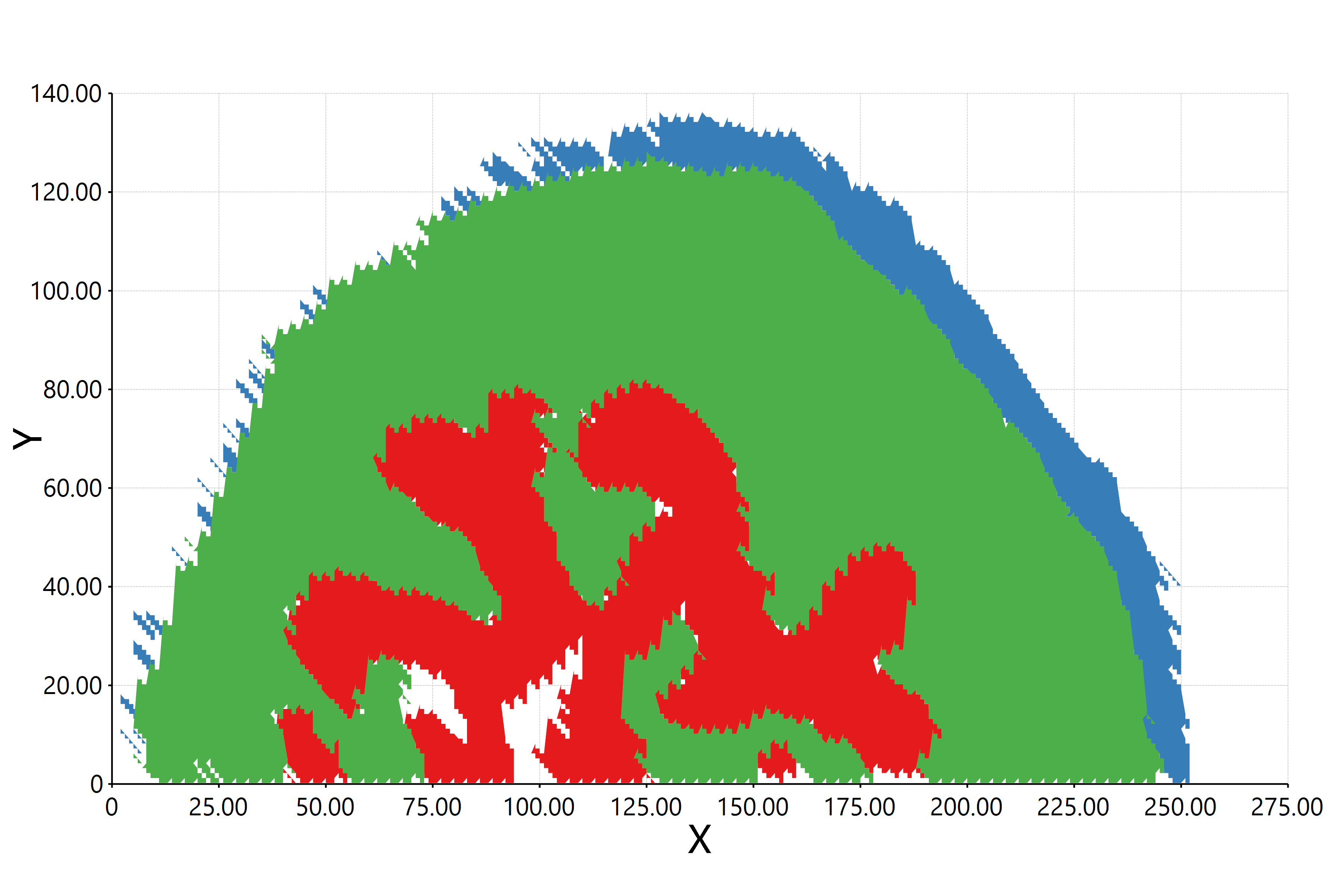
估计阅读时长: 10 分钟https://github.com/xieguigang/sciBASIC 最近在研究实现空间代谢组学中的一些特征区域的自动化划分分割。在得到了特征点集合之后,我们需要根据一些图像处理算法进行特征区域的提取操作。之前,我们尝试过基于绘制等高线图Marching Squares算法的方式来将特征点集合自动转换为特征区域的多边形,实现轮廓扫描获取的功能。但是实现的效果嘛,和实际的区域存在着一些较大的差异。 Order by Date Name Attachments HR2MSI mouse urinary bladder S096 - spatial regions […]
估计阅读时长: 2 分钟https://github.com/dotvanilla/vanilla 在Vanilla编译器项目之中,会需要一个程序模块将VisualBasic代码进行解析为语法树。然后我们基于此语法树就可以将VisualBasic项目转换为WAST源代码,从而实现编译为WebAssembly程序了。在这个步骤之中,我们可以通过一个微软官方的Roslyn编译器平台来实现。 Order by Date Name Attachments Roslyn-nuget • 107 kB • 274 click 2021年7月24日what-is-visual-studio • […]
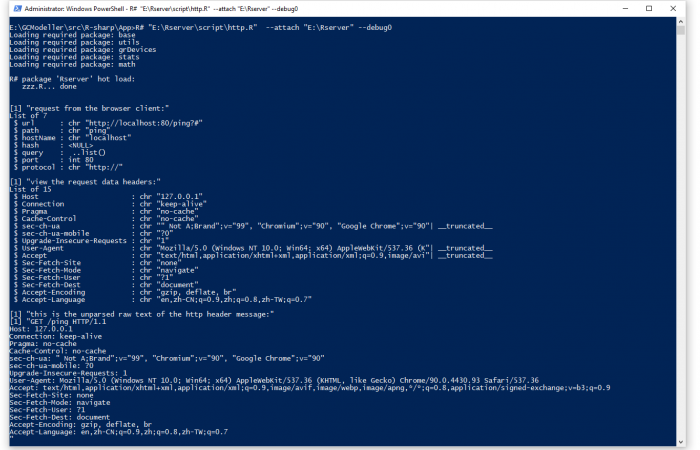
估计阅读时长: 5 分钟https://github.com/rsharp-lang/Rserver 在R语言之中,存在有一个FastRWeb的框架可以将R语言编写的脚本以http服务的方式运行于后台,供其他的语言进行调用。在R#语言之中,我也模仿着R语言之中的FastRWeb框架,创建了一个用于R#语言的web服务的程序包框架。 Order by Date Name Attachments httpr_commandline • 28 kB • 247 click 2021年6月16日http_PUT_test • […]
估计阅读时长: 12 分钟https://github.com/biocad-cloud/web HTTP协议(Hypertext Transfer Protocol)是建立在TCP协议基础上的一种文件传输协议。 Order by Date Name Attachments https-secure-webpages_waifu2x_art_noise3_scale_tta_1 • 770 kB • 242 click […]
估计阅读时长: 6 分钟今天在这里和大家聊一下《生物化学系统的计算分析》这本书,生物化学系统的计算分析这本书可以说得上是我的生物信息学学习生涯的启蒙书。 Order by Date Name Attachments Computational Analysis of Biochemical Systems • 875 kB • 225 […]
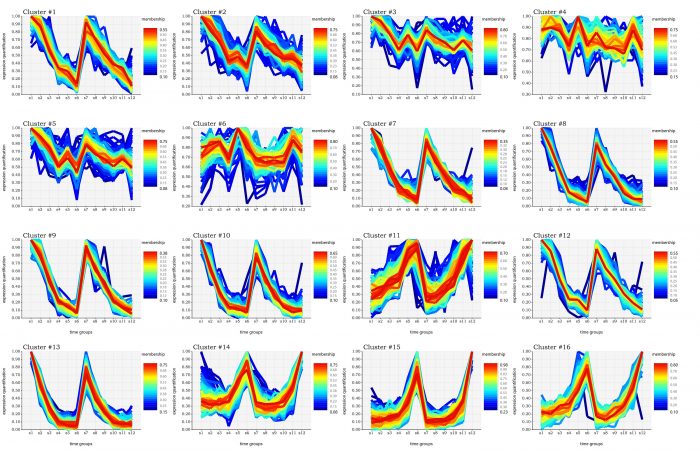
估计阅读时长: 11 分钟https://github.com/SMRUCC/GCModeller 在R语言之中,存在着一个用于进行表达数据的时间序列分析的程序包:TCseq。TCseq的全称为Time course sequencing,即时间序列分析,通过对表达矩阵进行时间上的模糊CMeans聚类,得到表达变化趋势一致的基因列表,进行基因表达的时间趋势分析。 在GCModeller之中,我仿照着TCseq程序包,自己编写了一个时间序列的聚类与可视化分析的R#程序包模块,在这里介绍给大家。 Order by Date Name Attachments Gene expression pattern visualization • 2 […]







Hello There. I found your website through the usage of msn. This is a very smartly written article. I will…
[…] […]
[…] […]
[…] 《为大语言模型运行添加工具调用》 […]
[…] 《从头创建一个DeepSeek客户端》 […]