文章阅读目录大纲
 https://github.com/rsharp-lang/bing-academic
https://github.com/rsharp-lang/bing-academic
必应学术是由微软必应团队联合微软研究院打造的免费学术搜索产品。旨在为广大研究人员提供海量的学术资源,并提供智能的语义搜索服务。目前已涵盖多学科学术论文、国际会议、权威期刊、知名学者等方面。搜索位置:http://cn.bing.com/academic。
之前在工作上进行文献搜索都是在必应搜索引擎来完成的。在最近的工作中比较关注一些topic,自己想要整理出知识点网络。由于手动整理会非常麻烦的,所以在这里开发了一些用于自动化建立知识网络的GraphQuery脚本,分享出来给大家。
关于在R#环境之中使用GraphQuery进行网页数据查询,大家可以阅读之前的博客文章:《GraphQuery开发》
网络请求:HTTP get
为了完成脚本进行知识网络的自动化建立,我们就需要拥有一个最基础网络请求函数进行搜索引擎的查询。我在开发R#运行时环境的时候,同时也编写了一些配套的R#函数用于封装一些R#环境中的工具函数,组成R#的一些基础包。在这里,我们使用R#的基础包之中的REnv::getHtml
#' Http get html or from cache
#'
#' @param url the url of the resource data on the remote server
#' @param interval the time interval in seconds for sleep after
#' request data from the remote server.
#'
#' @details using of the http get function required of set options of
#' ``http.cache_dir`` at first!
#'
const getHtml as function(url, interval = 3) {
# finally read data from cache
readText(http_get(
url = url,
streamTo = function(url, cache_file) {
writeLines(con = cache_file) {
# request from remote server
# if the cache is not hit,
# and then write it to the cache repository
content(requests.get(url));
}
},
interval = 3,
filetype = "html")
)
;
}执行GraphQuery脚本
在拥有了最基础的网络数据请求函数之后,我们就可以依据url得到html文本。接下来就是对html文件执行GraphQuery得到有效信息了。执行一段GraphQuery查询脚本的整个过程也是非常的简单的。我们可以参考之前的教程,写出下面的查询执行代码即可:
imports "graphquery" from "webKit";
#' Query html document
#'
#' @description Parse the given html document text and
#' query of the html components as R object.
#'
#' @param html the html document text
#' @param queryName the relative file name of the
#' graphquery definition file.
#'
#' @return a list/vector of html document element query result output
#'
const html_query as function(html, queryName) {
const queryfile as string = system.file(queryName, package = "BingAcademic");
print("query of html components from query file:");
print(queryfile);
Html::parse(html, strip = TRUE)
|> query(
graphquery = queryfile
|> readText
|> parseQuery
,
stripHtml = TRUE
)
;
}在上面的查询函数代码之中,我们首先使用Html::parse将凌乱的HTML文本解析为DOM树;接着可以调用query函数,将所需要进行查询的文档源Html文档对象以及graphquery这两个参数传入query函数即可。
由于必应搜索的html页面是在后端通过模板渲染出来,并且传输经过了html页面压缩,所以我们直接用编辑器打开会十分的凌乱。但是没有关系,因为页面中存在这解析操作的一些必要的标记信息,所以通过graphquery脚本我们任然可以很轻松的对信息数据进行查询。
虽然经过html压缩的文本非常的杂乱。但是在文本中任然会存在着必要的一些css标记信息:例如id编号,class列表,html标签名等信息。GraphQuery查询的目标对象是html文本解析出来的DOM树,所以html压缩并不会影响GraphQuery的解析操作。
相比较于通过正则表达式进行解析,GraphQuery查询的优势就是在于结构清晰,不会受到html压缩的影响。而且,由于html文本是被解析为DOM树的,所以在GraphQuery之中可以利用DOM文档树中的上下文信息进行辅助解析,对上下文信息的利用这个是正则表达式所无法实现的。
对于正则表达式解析而言,由于html文本在处于格式化或者压缩这两种状态的时候,html标签元素之间的空格和换行符可能会影响正则表达式的解析结果;并且html的标签大小写也可能会影响正则表达式的解析结果(例如没有处理好大小写的问题,可能有些标签就没有办法被匹配上了)。所以一般一种网页格式会需要对应编写一套正则表达式来做解析。但是对于GraphQuery而言,因为DOM树建立的时候,所有的节点标签名已经被归一化为统一的大小写格式,而空格换行等符号在DOM树中是不存在了的。所以无论目标html文档是出于任何状态都不会对GraphQuery的查询结果产生影响。
对于诸如解析出第二个被嵌套的DIV标签这样的具有上下文描述信息的数据,使用正则表达式会有一些麻烦。但是对于GraphQuery而言,由于其查询的对象为DOM树,所以处理这些具有上下文信息的数据解析,GraphQuery会更加方便。
编写GraphQuery脚本解析必应学术搜索结果
通过一段简单的查询脚本,就可以从凌乱的原始数据中得到非常明晰结构化的信息数据,这个就是GraphQuery查询的强大之处了。下面我们来学习怎样编写GraphQuery来从必应学术搜索结果页面提取文献信息吧。
我们首先来观察搜索结果页面中我们可以提取出哪些信息吧。从一个搜索结果页面上,最重要的信息就是页面上的结果列表了。虽然我们直接查看源代码是看到的一个经过html压缩的页面,但是好在有谷歌浏览器的开发者工具这个神器。我们可以很轻松的看到搜索结果列表中结果的html DOM文档树的结构信息。
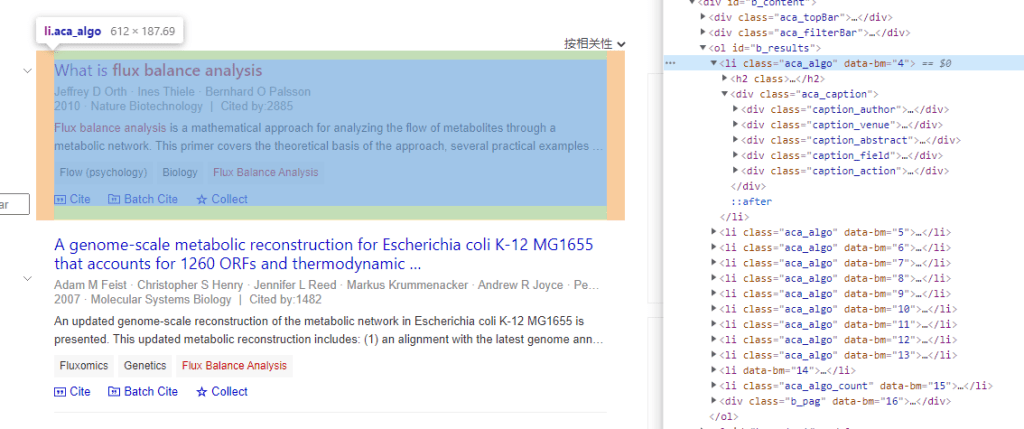
那,从上面的截图可以看得出来,我们只需要解析class为aca_algo的列表元素即可得到结果列表。所以在我们的graphquery脚本之中,最基础的查询就是列表元素查询,通过观察页面,得到如下所示的查询结果数据结构:
list css(".aca_algo", "*") [
{
title
authors
publication
abstract
fields
}
]接下来我们就需要按照对应DOM文档树结构,往里面填充查询表达式即可。
查询实例教程
注:其实在这里我并非想要教会大家如何解析必应学术搜索的页面解析。而是想要大家可以通过这个解析的实例来了解GraphQuery查询引擎的工作原理,以及如何编写查询表达式来处理大家在工作中遇到的实际问题。
title元素

可以看得到,title字段的值,是位于一个h2标签内。所以我们对h2标签取文本值可以得到标题文本。文章在必应搜索数据库中的唯一编号在a标签的链接属性中,我们取出链接href属性,然后基于正则文本解析,得到id的值即可:
title css("h2", 0) {
guid css("a")
| attr("href")
| regexp("id[=][a-z0-9]+")
| tagValue("=", "value")
title text() | strip() | trim()
}作者列表

至于文章的作者列表,是位于caption_author类的div标签中的a标签列表中。我们可以通过数组来存储这个列表数据。对于每一个作者而言,其包含有作者名字,以及作者在必应数据库中的唯一编号这两个属性,也可以按照文章title类似的方法解析出来:
authors css(".aca_caption", 0)
| css(".caption_author", 0)
| css("a")
[
{
id attr("href")
| regexp("id[=]\d+")
| tagValue("=", "value")
name text() | strip()
}
]出版信息以及引用次数

对于出版信息这一个字段的信息的解析,要稍微复杂一些。但是我们稍作观察,就可以看得到:出版信息和引用次数之间是通过一个竖线符号分割开的。所以我们可以先将文本全部解析出来,然后再做字符串分割即可。还记得前面的例子中有一个tagValue函数么?我们在这里就使用这个函数来做分割:按照竖线符号分割,得到name和value部分构成一个tag value对。name部分就是出版物信息,而value部分就是引用次数啦。我们再对引用次数信息替换一下Cites:
publication css(".caption_venue", 0) {
ref text()
| trim()
| tagValue("|", "name")
| strip()
cites text()
| trim()
| tagValue("|", "value")
| replace("Cited by:", "")
| trim()
}摘要文本

这个就非常简单了,直接取出文本数据,然后trim一下首尾出现的空白以及换行即可。
abstract css(".caption_abstract", 0) | text() | strip()关键词列表

对于关键词列表,其结构和作者列表是一样的,按照相同的方法取出数据:
fields css(".caption_field", 0) | css("a") [
{
id attr("href")
| regexp("id[=]\d+")
| tagValue("=", "value")
term text() | strip()
}
]好了,现在所有的数据查询都齐全了。上面的完整实例查询,大家可以进入项目的代码库中查看这个查询表达式文件内容:【graphquery/listPage.graphquery】。
查询结果展示
将上面所提到的函数和查询脚本串接起来,执行一下,就可以得到一个必应学术的查询过程。在R#环境中执行下面的查询函数,就可以得到查询结果:
imports ["Html", "http", "graphquery"] from "webKit";
#' Do bing academic search
#'
#' @param term the term string to be searched
#'
#' @return a list of term search result which is query from
#' the bing academic search engine.
#'
const search as function(term) {
const urlq as string = `https://cn.bing.com/academic/search?q=${urlencode(term)}`;
const html as string = REnv::getHtml(urlq);
const result = html
|> BingAcademic::html_query("graphquery/listPage.graphquery")
|> sapply(function(i) {
.summary(
title = i$title$title,
guid = i$title$guid,
ref = i$publication$ref,
cites = i$publication$cites,
abstract = i$abstract,
authors = frameData(i$authors),
fields = frameData(i$fields, "term")
);
})
;
result;
}好的,可以看到,查询的结果完全OK!

- 基因组功能注释(EC Number)的向量化嵌入 - 2026年2月11日
- TF-IDF与N-gram One-hot文档嵌入算法原理 - 2026年2月10日
- CenterStar多序列比对算法 - 2026年1月8日



2 Responses
[…] FBA分析的一些相关知识库信息的建立,大家可以阅读之前写的两篇博客文章:《数据可视化】基于必应学术搜索的知识网络可视化》和《GraphQuery】使用GraphQuery查询必应学术》 […]
[…] 【GraphQuery】使用GraphQuery查询必应学术 […]