估计阅读时长: 15 分钟https://gcmodeller.org 在这篇博客文章之中,我主要是来详细介绍一下是如何从头开始实现Phenograph单细胞分型算法的。在之前的一篇博客文章《【单细胞组学】PhenoGraph单细胞分型》之中,我们介绍了Phenograph算法的简单原理,以及一个在R语言之中所实现的Phenograph算法的程序包Rphenograph。在这里我主要是详细介绍在GCModeller软件之中所实现的VisualBasic语言版本的Phenograph单细胞分型算法。 Attachments Rphenograph • 236 kB • 658 click 2021年9月20日
估计阅读时长: 17 分钟https://github.com/xieguigang/sciBASIC/tree/master/gr/Microsoft.VisualBasic.Imaging/Drawing3D 因为大家大多数都是从小接受电子游戏,所以长大了之后能够自己从零开始开发一个完整的3维图形引擎是每一个男程序员的梦想。这个就像玩机械的男人的梦想就是自己从头开始组装一辆汽车。还好这个梦想我在几年前就已经实现了。 Order by Date Name Attachments Cube3D_VB.NET • 4 MB • 739 click 2021年9月19日Cube_screenshot • […]
估计阅读时长: 6 分钟之前在阅读一个使用rust语言编写的contour tracing算法模块的源代码的时候,其中有一个向量的左旋以及右旋的操作。这个操作的具体的含义是和在算法中的轮廓边缘像素的读取方向有关:因为访问方向是一个二维平面的概念,但是在代码中我们只能够使用一个一维的数组的来存储这个二维的信息。所以在这段rust代码之中,作者很巧妙的使用了向量的左旋以及右旋操作来实现一维数组中对二维平面上的方位的访问操作。 Order by Date Name Attachments RotateVector • 30 kB • 674 click 2021年9月16日Full • […]
估计阅读时长: 11 分钟https://github.com/xieguigang/sciBASIC 最近在研究实现空间代谢组学中的一些特征区域的自动化划分分割。在得到了特征点集合之后,我们需要根据一些图像处理算法进行特征区域的提取操作。之前,我们尝试过基于绘制等高线图Marching Squares算法的方式来将特征点集合自动转换为特征区域的多边形,实现轮廓扫描获取的功能。但是实现的效果嘛,和实际的区域存在着一些较大的差异。 Order by Date Name Attachments HR2MSI mouse urinary bladder S096 - spatial regions […]
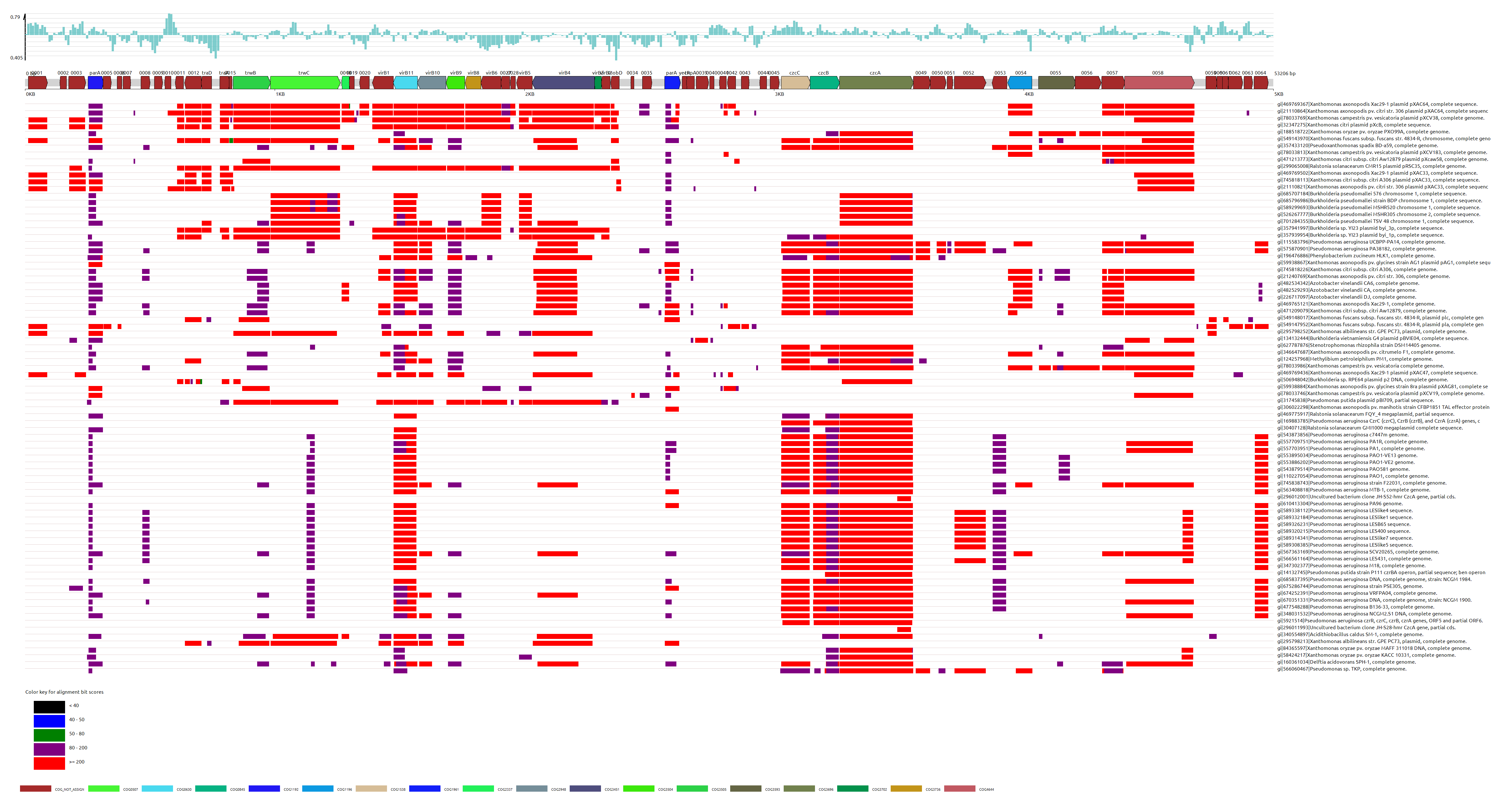
估计阅读时长: 14 分钟在基因组学研究中,将新测序的基因或者针对目标基因组进行基于KEGG代谢通路体系的虚拟细胞建模,都会需要将目标基因组与已知功能基因进行比对注释。KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库通过其KEGG Orthology (KO)系统,为基因功能注释提供了一个强大的平台。KO系统将功能上保守的直系同源基因归为一类,每个KO条目(K编号)代表一个直系同源基因群,这些基因在不同物种中通常执行相似的生物学功能。因此,将新基因的序列与KEGG数据库中的已知基因进行比对,可以推断其可能的KO编号,从而将其功能映射到KEGG通路图或功能层级中。 Order by Date Name Attachments kegg_overview • 313 […]
估计阅读时长: 15 分钟https://github.com/xieguigang/sciBASIC 最近在空间代谢组学中的质谱成像应用开发过程中,会需要使用到一些图像处理算法对原始的质谱成像结果图片进行诸如平滑,放大等处理。顺着图像平滑的算法搜索,通过搜索引擎找到了一个年代比较久远的图像处理算法博客文章,将其中的图像算法重新实现了一下,在这里分享给大家。 Order by Date Name Attachments lena • 558 kB • 632 click 2021年9月10日lenalena • […]
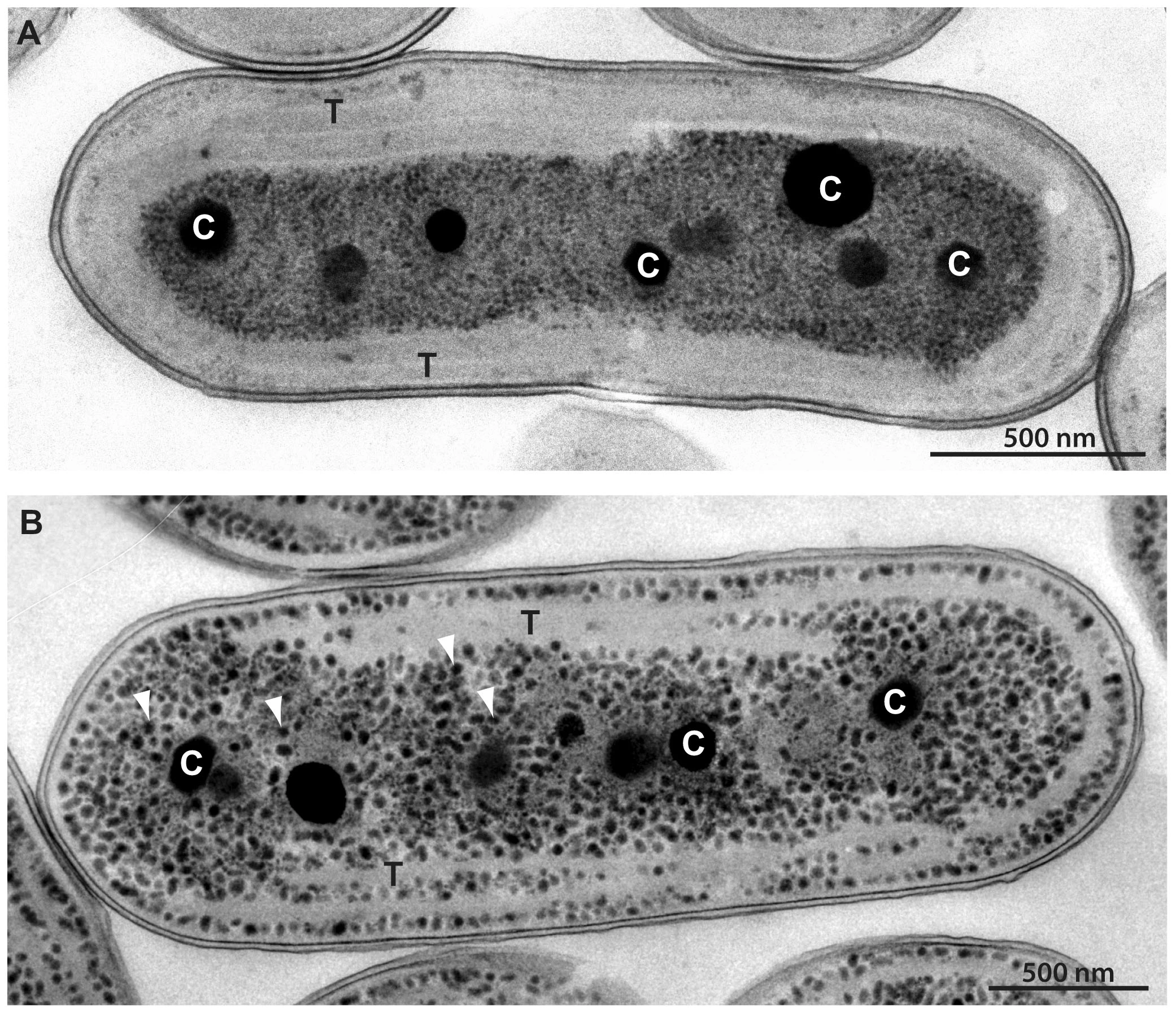
估计阅读时长: 10 分钟https://gcmodeller.org/ 流平衡分析(flux balance analysis)是一种可以用来构建和模拟分析基因组级别的代谢网络的数学方法。流平衡分析是系统生物学(system biology)的一个重要的分析手段。不同于以湿实验的代谢通量分析(metabolic flux analysis, MFA),FBA是用数学方法对代谢网络里的代谢流进行拟合分析。 Order by Date Name Attachments Electron micrographs of […]
估计阅读时长: 9 分钟https://github.com/xieguigang/sciBASIC 在实际应用的机器学习方法里,GradientTree Boosting (GBDT)是一个在很多应用里都很出彩的技术。XGBoost是一套提升树可扩展的机器学习系统。XGBoost全名叫(eXtreme Gradient Boosting)极端梯度提升。它是大规模并行boosted tree的工具,XGBoost 所应用的算法就是 GBDT(gradient boosting decision tree)的改进,既可以用于分类也可以用于回归问题中。 Order by Date Name […]

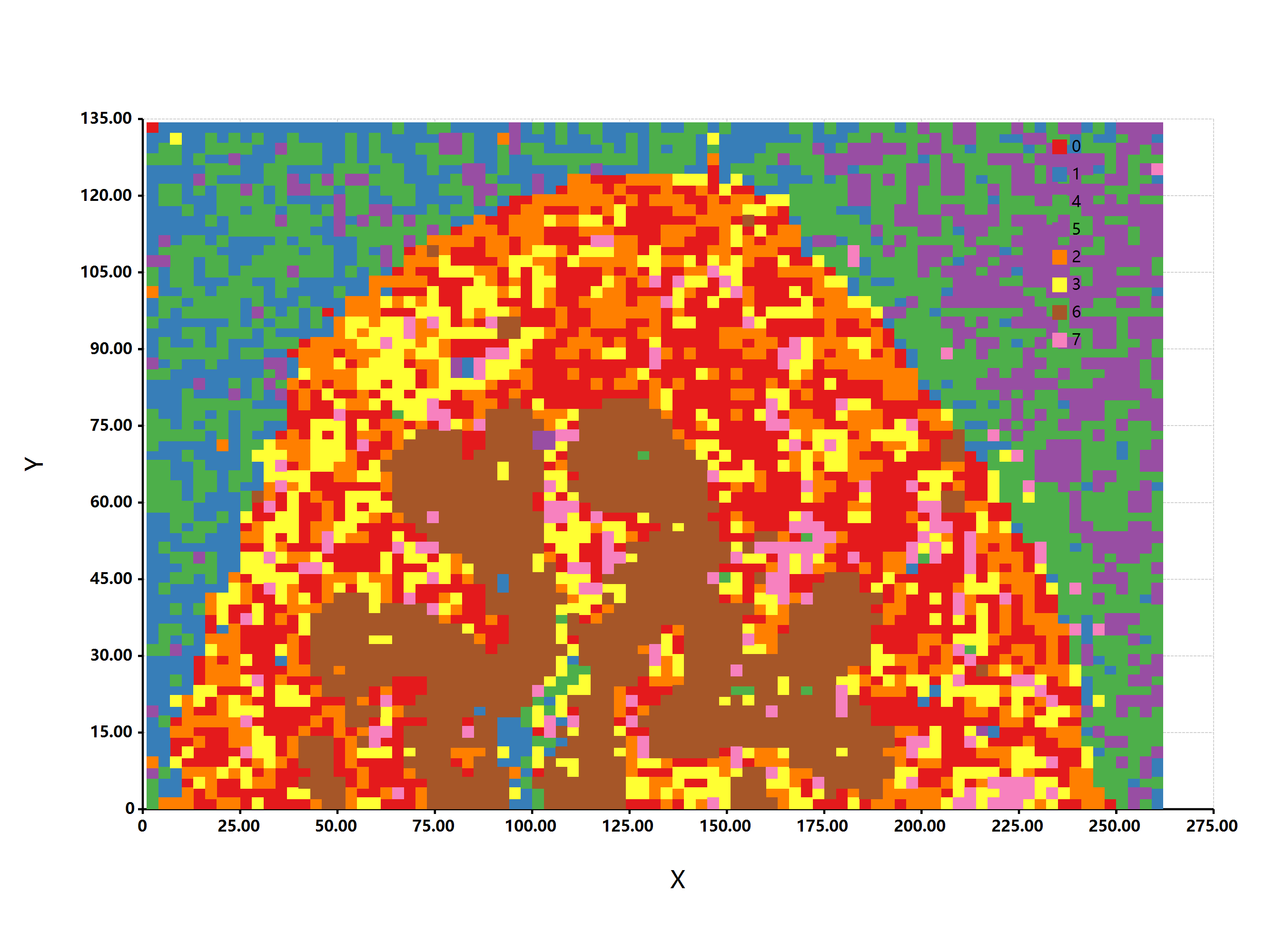
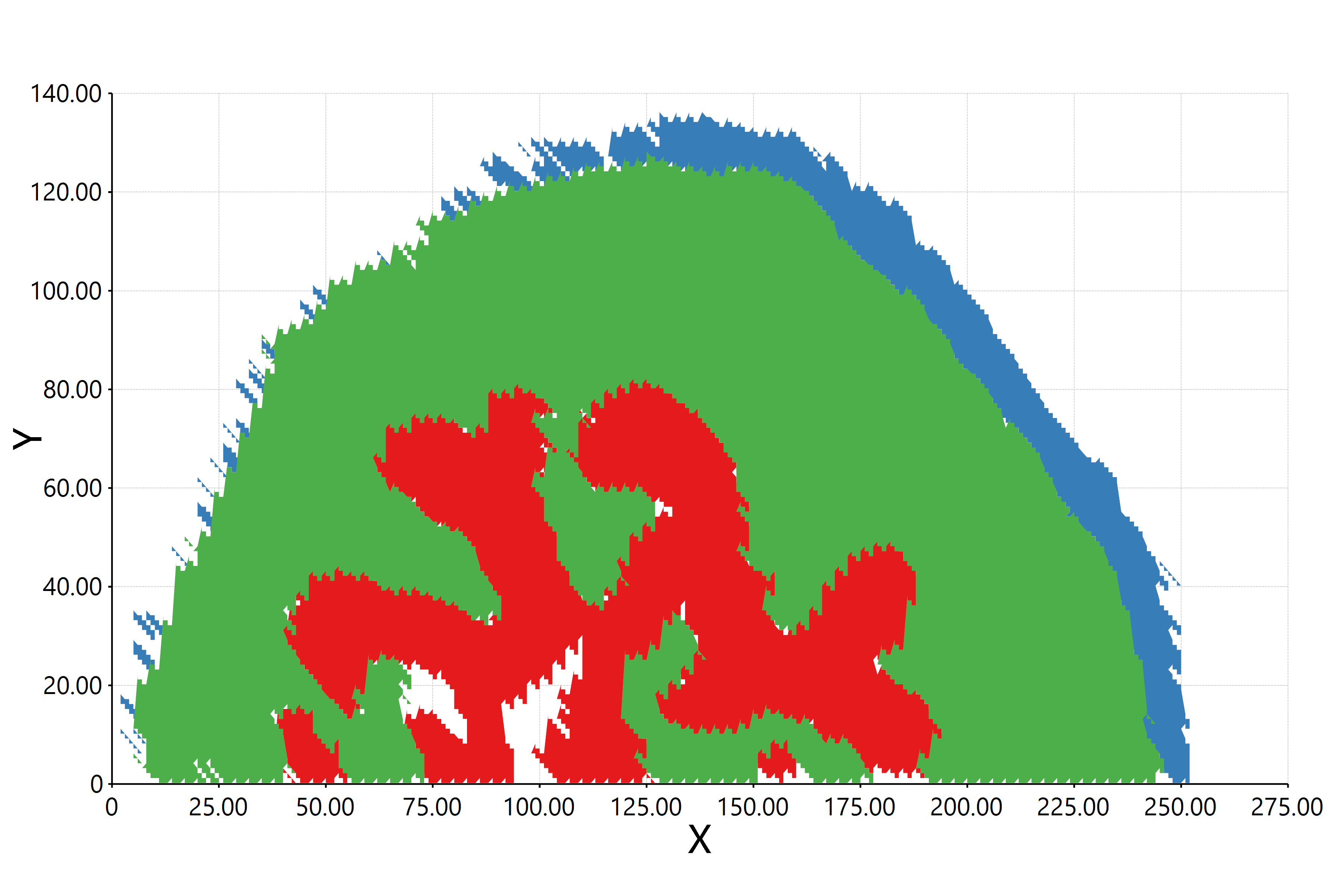

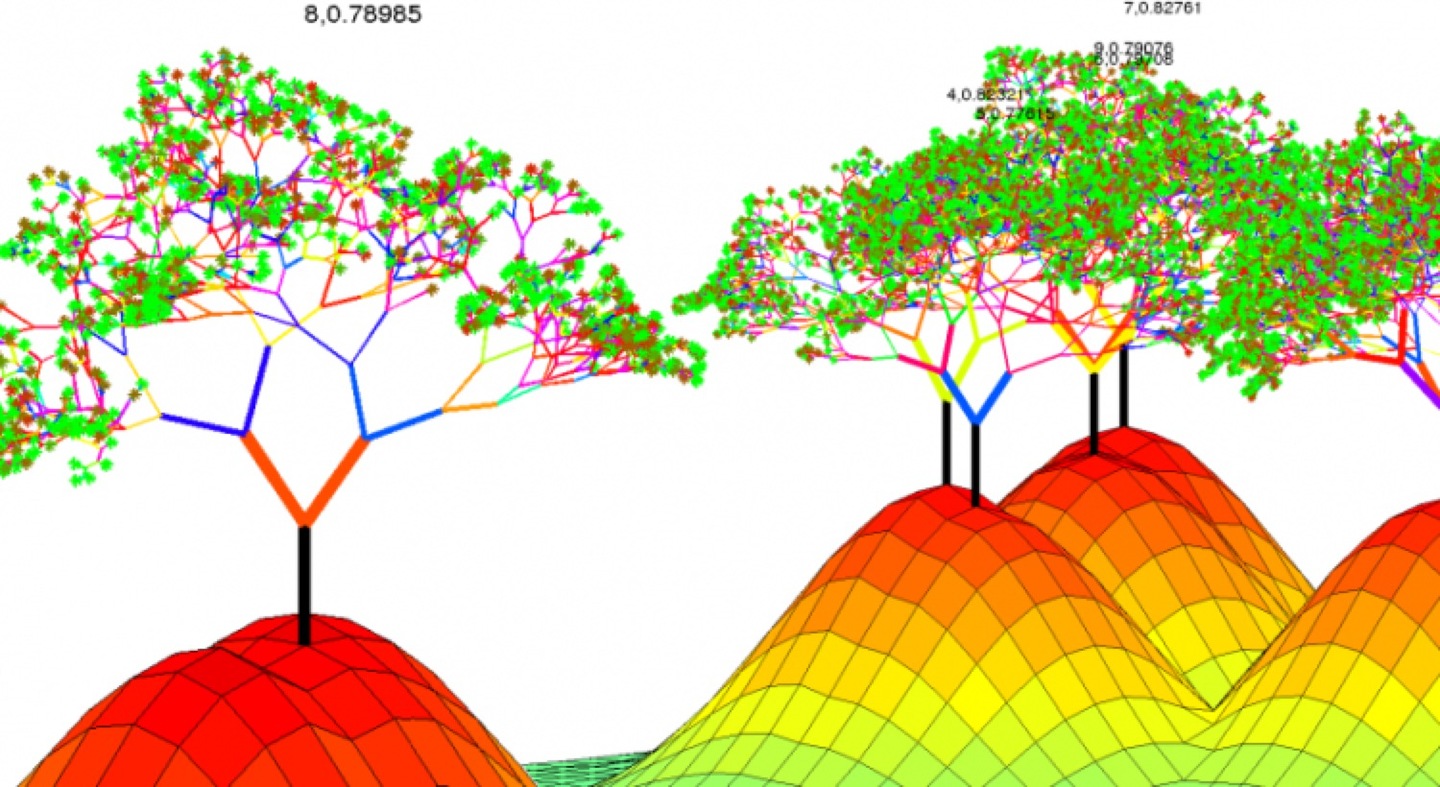

[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]