估计阅读时长: 5 分钟单细胞轨迹可以揭示基因调控如何控制细胞命运:大多数细胞状态转变,无论是在发育,重编程或者是疾病异常状态,都以基因表达变化的级联为特征。 Order by Date Name Attachments vec • 722 kB • 674 click 2022年3月17日Slide10 • 14 […]
估计阅读时长: 5 分钟https://github.com/xieguigang/graphQL 构建一个图数据库,可以用来帮我们解决复杂的知识关联计算问题。例如我们想要程序向我们回答dihydrogen oxide与water是否是同一个东西。如果光从字符串比较角度上面来看待这个问题的话,很显然,二者的字符串比较结果肯定是False。面对上面的这个问题,图数据库则可以很简单的向我们回答道上面的两个字符串都是指代的同一个东西。 Order by Date Name Attachments tumblr_inline_mqvdlydGCp1qz4rgp • 124 kB • 610 click 2022年3月5日Capture […]
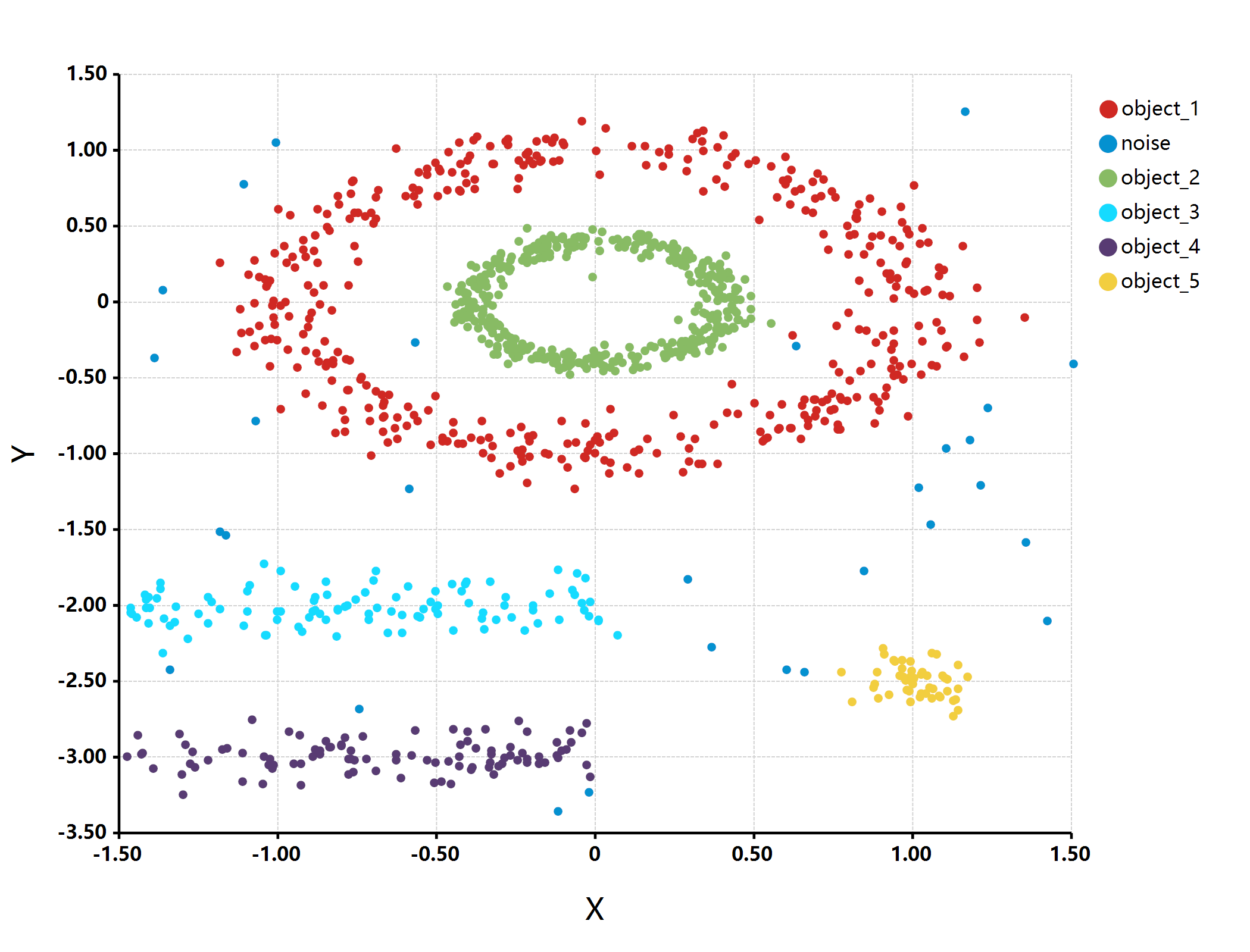
估计阅读时长: 2 分钟imports "clustering" from "MLkit"; require(graphics2D); multishapes = read.csv("./multishapes.csv"); [x, y] = list(multishapes[, "x"], multishapes[, "y"]); print(multishapes, […]
估计阅读时长: 18 分钟https://github.com/rsharp-lang/R-sharp/tree/master/studio/RData 如果我们需要将上游的R数据分析环境之中的数据集串流至下游的R#数据分析环境之中,构建出一个不同的数据分析环境混合在一块的自动化数据分析流程。我们一般会需要将上游的R环境之中的数据符号对象以RData的格式串流到下游环境中,下游环境进行反序列化加载数据到环境中执行相应的分析。例如在下游执行定制化程度更高的数据作图,将数据以在上游R环境中比较困难实现的其他二进制文件格式进行保存,或者进行分布式的跨物理机的集群化计算,等等用于实现单纯依靠R环境所比较困难实现的功能。 从上一篇博客文章之中我们比较下详细的了解了RData数据文件的文件格式以及对应的读取操作。在这篇文章之中我们来了解如何基于我们通过对RData文件读取操作所获取得到的链表数据进行反序列化操作,将R环境之中的数据集串流加载到下游的R#数据分析环境之中。 Order by Date Name Attachments rstudio-og-fb-1-1024x538 • 39 kB • 672 click 2021年12月4日read-vector […]
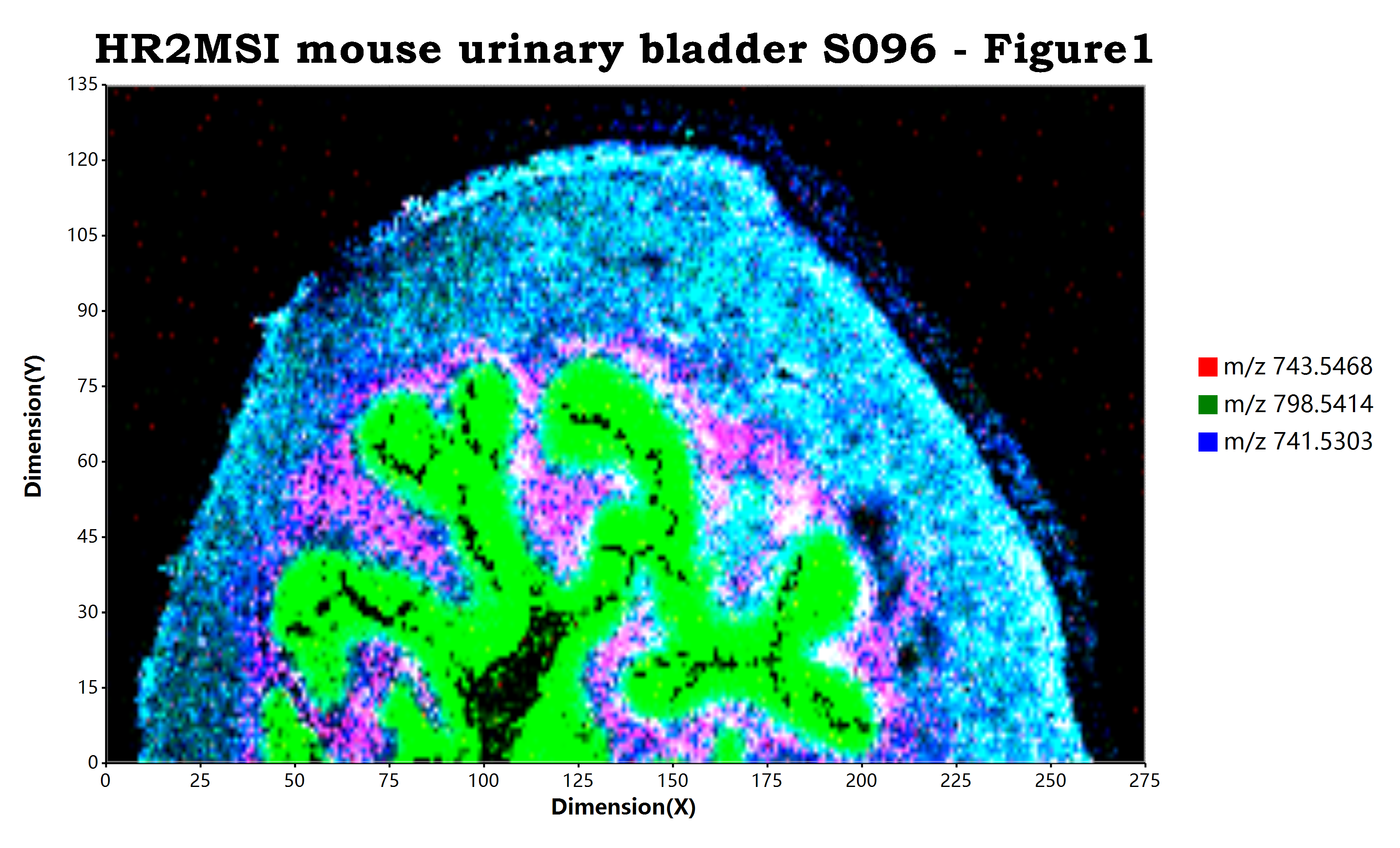
估计阅读时长: 10 分钟https://github.com/xieguigang/ms-imaging Order by Date Name Attachments HR2MSI_mouse_urinary_bladder_S096_RGB • 7 MB • 691 click 2021年11月13日peerj-cs-07-585 • 16 […]
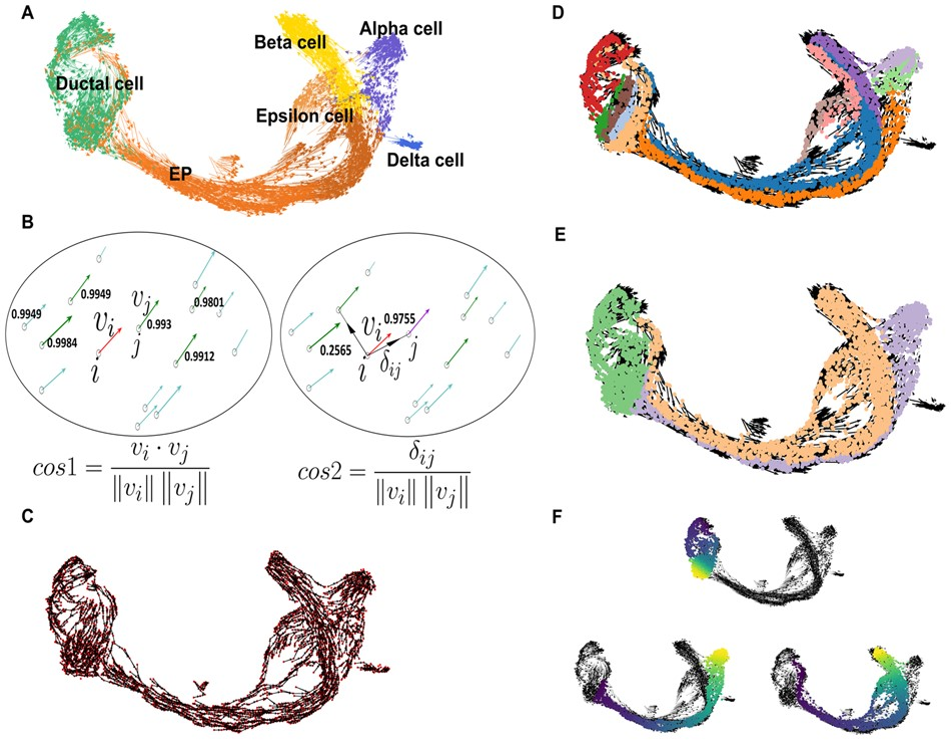
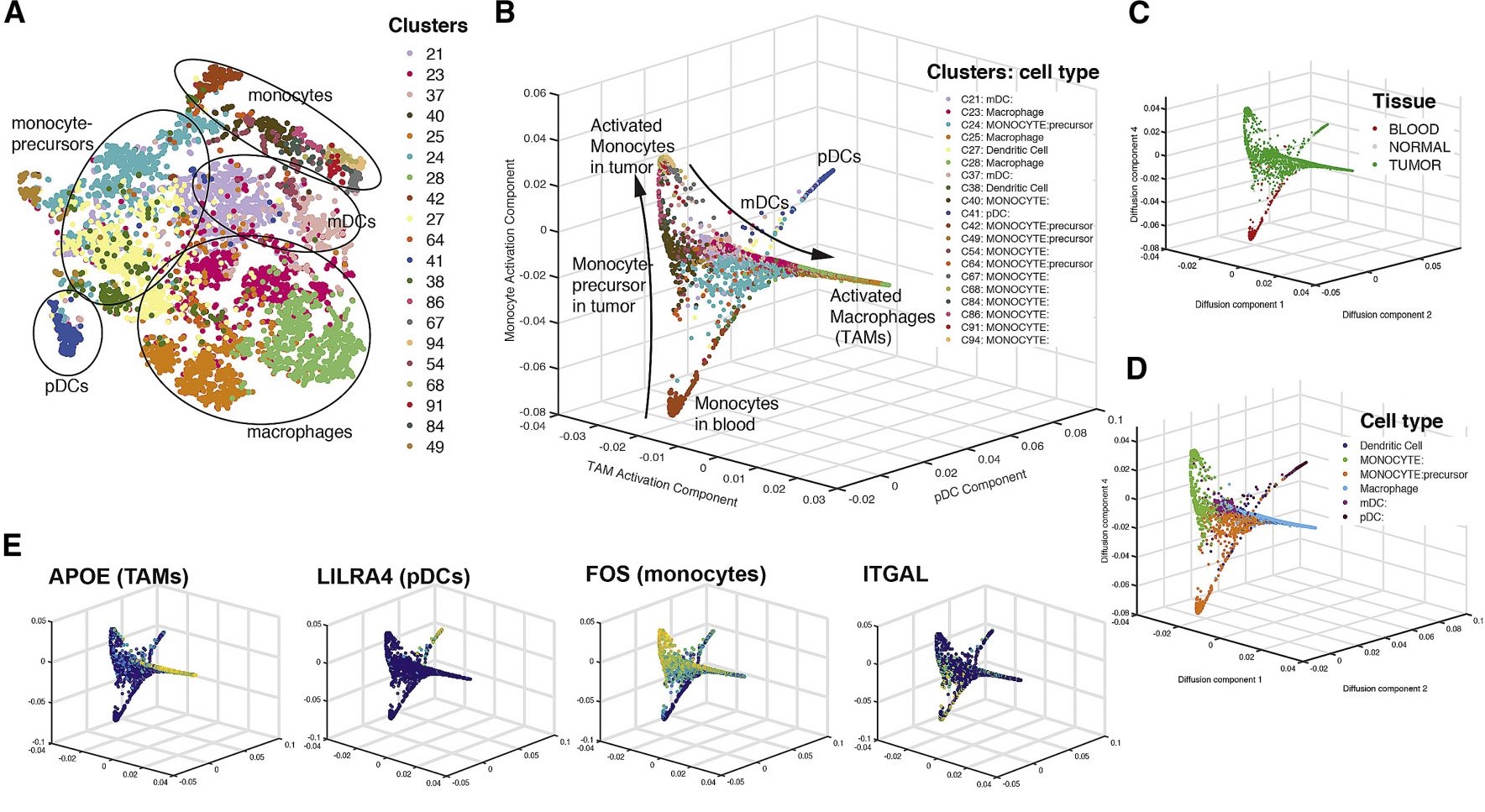
估计阅读时长: 14 分钟https://github.com/rsharp-lang/ggplot 之前在阅读一篇单细胞组学数据分析的文献,觉得在文献之中有一些三维散点图用于展示降维聚类结果的效果非常的好看。于是自己在R#语言之中的ggplot程序包的2D绘图的功能基础之上,进行了三维图形数据可视化功能的开发。 (A) t-SNE map projecting myeloid cells from BC1-8 patients (all tissues). Cells are colored […]
估计阅读时长: 11 分钟https://github.com/xieguigang/ms-imaging Mass spectrometry imaging ( MSI) is a technique used in mass spectrometry to visualize the […]
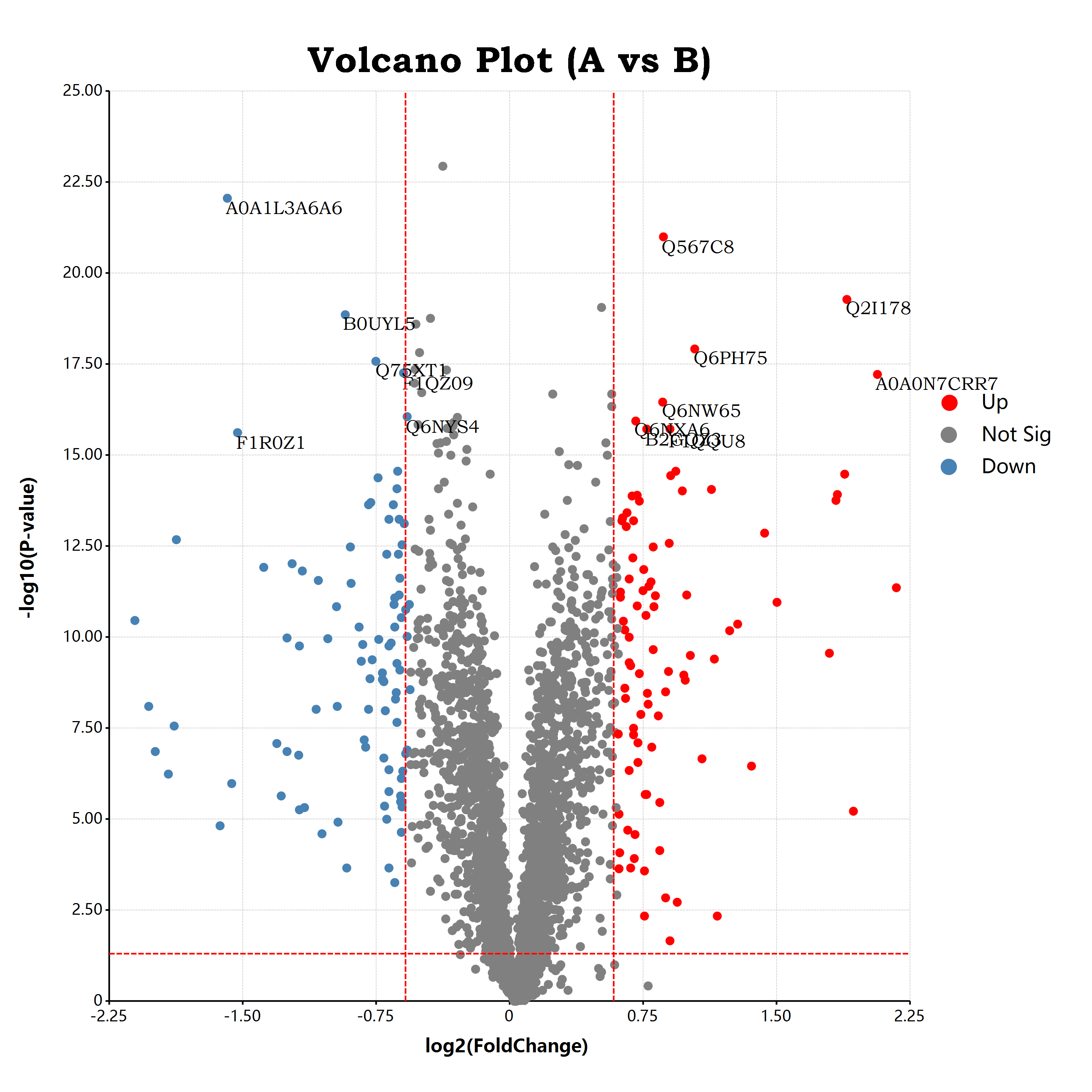
估计阅读时长: 17 分钟https://github.com/rsharp-lang/ggplot 接上一篇博客文章中谈到,我们已经通过R#语言之中的ggplot程序包绘制出了一个可以使用的火山图。在这里,我们将会通过在火山图上添加更多的可视化元素来为大家介绍R#语言之中的ggplot程序包的进阶使用方式。 Order by Date Name Attachments volcano • 651 kB • 728 click 2021年10月9日volcano • […]
估计阅读时长: 11 分钟https://github.com/rsharp-lang/ggplot 在生物信息学中的组学数据分析领域内,有一个非常常见的数据可视化图表:应用于可视化两两组别比对结果的火山图。在火山图之中,X坐标轴一般是log2FC,纵坐标Y轴,则一般是t检验的pvalue的-log10转换之后的值。由于fold change有大于1的值,A/B大于1,表示A的表达量高于B的表达量,反之小于一表示A的表达量低于B的表达量。这样子fold change经过log2转换之后,就会出现负数,散点一般呈轴对称分布在X=0的位置周围。这样子绘制出来的散点图就有点类似于火山喷发的样子了。 Order by Date Name Attachments a679af1eb9ffbfbad48c18d563ea51f3 • 45 kB • 700 click […]
估计阅读时长: 7 分钟https://github.com/rsharp-lang/ggplot 一张统计图形就是从数据到几何对象(geometric object, 缩写为geom, 包括点、线、条形等)的图形属性(aesthetic attributes, 缩写为aes, 包括颜色、形状、大小等)的一个映射。此外, 图形中还可能包含数据的统计变换(statistical transformation, 缩写为stats), 最后绘制在某个特定的坐标系(coordinate system, 缩写为coord)中, 而分面(facet, 指将绘图窗口划分为若干个子窗口)则可以用来生成数据中不同子集的图形。 […]




[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]