文章阅读目录大纲
 https://github.com/rsharp-lang/ggplot
https://github.com/rsharp-lang/ggplot
在完成了前面所提到的ANOVA检验模块的代码开发编写工作之后,之前一直悬在我心里面的完善R#
目前在ggplot程序包之中完成的主要统计图表多是应用于组学数据分析之中,例如火山散点图,柱状图,条形图,小提琴图等。下面我就来一一介绍在R#
火山图绘制

# create ggplot layers and tweaks via ggplot style options
ggplot(volcano, aes(x = "log2(FC)", y = "p.value"), padding = "padding:250px 500px 250px 300px;")
+ geom_point(aes(color = "factor"), color = "black", shape = "circle", size = 50,alpha = 0.7)
+ scale_colour_manual(values = list(
Up = "#D22628",
"Not Sig" = "black",
Down = "#0091D5"
), alpha = 0.7)
+ geom_text(aes(label = "ID"), check_overlap = TRUE, size = 8)
+ geom_hline(yintercept = -log10(0.05), color = "red", line.width = 5, linetype = "dash")
+ geom_vline(xintercept = log2(foldchange), color = "red", line.width = 5, linetype = "dash")
+ geom_vline(xintercept = -log2(foldchange), color = "red", line.width = 5, linetype = "dash")
+ labs(x = "log2(FoldChange)", y = "-log10(P-value)")
+ ggtitle("Volcano Plot (A vs B)")
+ scale_x_continuous(labels = "F2")
+ scale_y_continuous(labels = "F2")
+ theme(plot.title = element_text(family = "Cambria Math", size = 20))
;多组比较数据可视化
为什么在最开始提到在R#
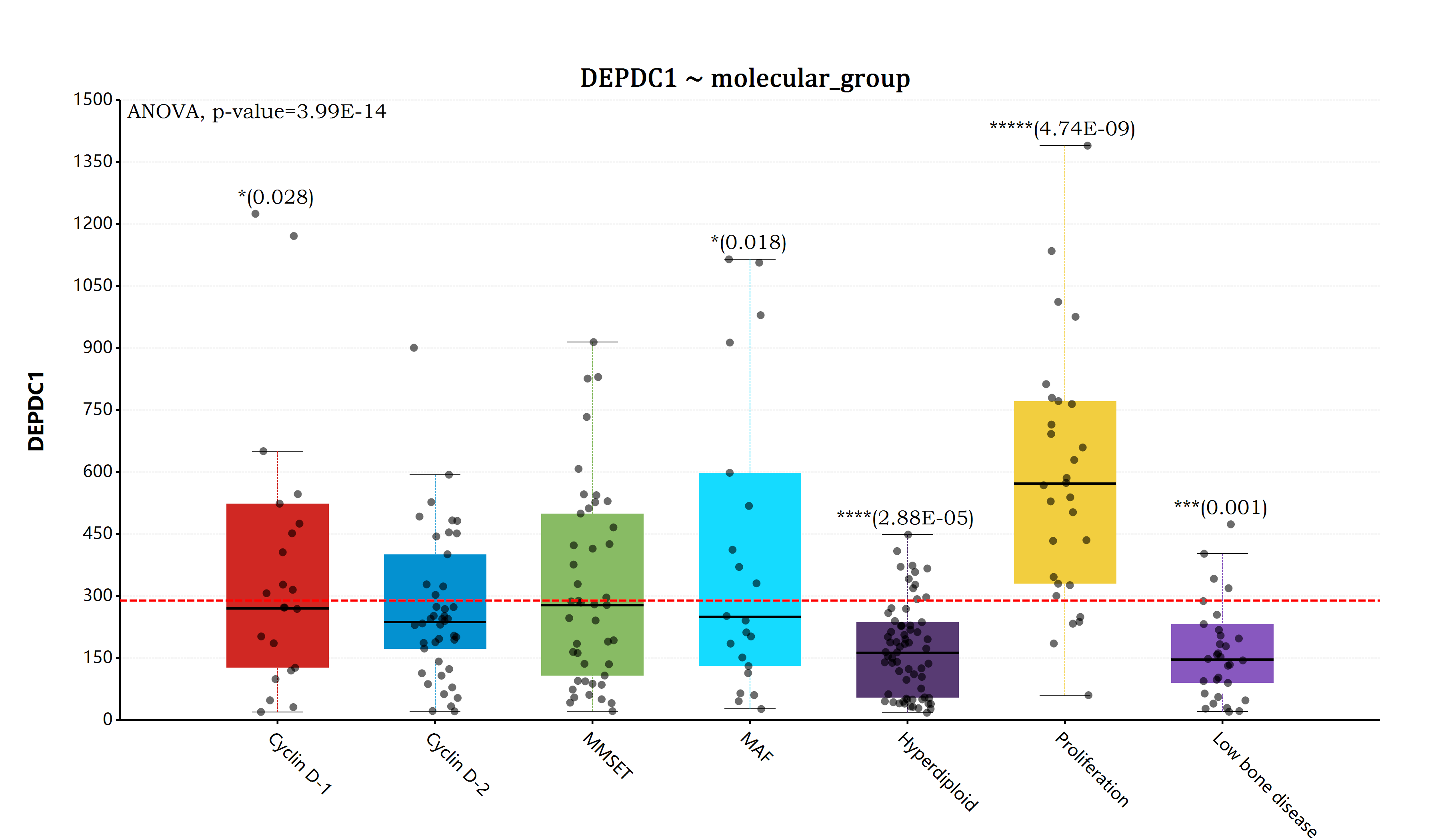
但是好在昨天下定了决心,一鼓作气将ANOVA检验分析的原理从头吃到了尾,在完全理解透了F检验和相应的Pvalue计算过程之后,将ANOVA检验分析的代码模块给成功从头实现了出来。现在我可以在ggplot程序包之中内置对应的统计学检验分析元素,这样子就可以绘制出很完美的多组比较结果的统计图了:



上面的三幅图的绘制代码非常的简单,大家只需要将R语言之中的ggplot2程序包的绘图代码复制出来,R#
ggplot(myeloma, aes(x = "molecular_group", y = "DEPDC1"))
+ geom_violin(width = 0.65)
+ geom_jitter(width = 0.3)
# Add horizontal line at base mean
+ geom_hline(yintercept = mean(myeloma$DEPDC1), linetype="dash", line.width = 6, color = "red")
+ ggtitle("DEPDC1 ~ molecular_group")
+ ylab("DEPDC1")
+ xlab("")
+ scale_y_continuous(labels = "F0")
# Add global annova p-value
+ stat_compare_means(method = "anova", label.y = 1600)
# Pairwise comparison against all
+ stat_compare_means(label = "p.signif", method = "t.test", ref.group = ".all.", hide.ns = TRUE)
+ theme(axis.text.x = element_text(angle = 45), plot.title = element_text(family = "Cambria Math", size = 16))
;对ggplot添加统计检验代码实现
从前面已经了解到ggplot程序包目前已经内置对统计检验的支持,可以直接调用对应的函数进行统计分析,那对应的功能是怎样实现的呢?实际上,在ggplot图表渲染过程之中,我们可以将统计检验的结果当作为相应的text图层。这样子我们就可以基于text图层的绘制代码,在完成了相应的统计检验分析之后就可以进行结果的可视化了。
和ggplot2程序包之中一致,在ggplot程序包之中,也是可以通过stat_compare_means
<ExportAPI("stat_compare_means")>
Public Function stat_compare_means(Optional method As String = "anova",
Optional ref_group As String = ".all.",
Optional hide_ns As Boolean = True) As ggplotLayer
Return New ggplotStatPvalue With {
.method = method,
.hide_ns = hide_ns,
.ref_group = ref_group
}
End Function上面的函数工作原理非常的容易理解,就是根据参数值创建一个对应的统计检验图层对象即可。
想了解关于ggplot的图层是如何工作的,大家可以阅读之前写的一篇博客文章:
【数据可视化】对ggplot程序包的从头实现
在R#
Public Class ggplotStatPvalue : Inherits ggplotGroup
Public Property method As String = "anova"
Public Property ref_group As String = ".all."
Public Property hide_ns As Boolean = True
Protected Overrides Function PlotOrdinal(stream As ggplotPipeline, x As OrdinalScale) As IggplotLegendElement
Select Case method.ToLower
Case "anova" : Call plotAnova(stream, x)
Case "t.test" : Call plotTtest(stream, x)
Case Else
Throw New NotImplementedException(method)
End Select
Return Nothing
End Function
End Class可以看得到,目前在程序包之中已经实现了两两比较的t检验以及多组之间进行比较计算的ANOVA检验方法。在检验分析的图层对象之中,程序代码会根据对应的method名称,调用执行对应的检验方法函数,下面来分别查看相应的检验分析函数的实现过程吧。
T检验
由于t检验是应用于两两比较上面的,所以我们首先需要有一个参考组。在ggplot之中,进行t检验计算的参考组可以有两种类型:所有组别的均值作为参考组或者某一个单独的组别的均值作为参考组。所以根据对应的描述,我们在这里保持和ggplot2程序包一致,将.all.
Private Sub plotTtest(stream As ggplotPipeline, xscale As OrdinalScale)
Dim data = getDataGroups(stream).ToArray
Dim ref As Double()
Dim font As Font = CSSFont.TryParse(stream.theme.tagCSS).GDIObject(stream.g.Dpi)
Dim lbsize As SizeF
If ref_group = ".all." Then
ref = data _
.Select(Function(v) v.value) _
.IteratesALL _
.ToArray
Else
ref = data _
.Where(Function(v) v.name = ref_group) _
.FirstOrDefault _
.value
If ref Is Nothing Then
Throw New EntryPointNotFoundException($"missing the reference group data: '{ref_group}'!")
End If
End If
' ...
End Sub那在得到了对应的参考组之后,我们就可以对所有的组别数据进行一次循环,进行两两比较计算分析了:
For Each group As NamedCollection(Of Double) In data
Dim p As TwoSampleResult = t.Test(group, ref, varEqual:=True)
Dim pvalue As Double = p.Pvalue
Dim sig As String
If pvalue <= 0.00001 Then
sig = $"*****({pvalue.ToString("G3")})"
ElseIf pvalue <= 0.0001 Then
sig = $"****({pvalue.ToString("G3")})"
ElseIf pvalue <= 0.001 Then
sig = $"***({pvalue.ToString("F3")})"
ElseIf pvalue <= 0.01 Then
sig = $"**({pvalue.ToString("F3")})"
ElseIf pvalue <= 0.05 Then
sig = $"*({pvalue.ToString("F3")})"
ElseIf pvalue <= 0.1 Then
sig = "."
ElseIf hide_ns Then
Continue For
Else
sig = $"n.sig"
End If
lbsize = stream.g.MeasureString(sig, font)
Dim x As Double = xscale(group.name) - lbsize.Width / 2
Dim y As Double = stream.scale.TranslateY(group.Max) - lbsize.Height * 1.125
Call stream.g.DrawString(sig, font, Brushes.Black, New PointF(x, y))
Next直接调用对应的t检验函数t.test即可,上面所实现的t检验的功能在ggplot程序包之中的调用方式为:
# Pairwise comparison against all
stat_compare_means(label = "p.signif", method = "t.test", ref.group = ".all.", hide.ns = TRUE)多组ANOVA检验
前面所看到的是ggplot程序包之中对t检验的代码实现,那现在要讲解的就是多组数据比对所使用到的ANOVA检验分析的实现:
Private Sub plotAnova(stream As ggplotPipeline, x As OrdinalScale)
Dim data = getDataGroups(stream).ToArray
Dim observations As Double()() = data _
.Select(Function(v) v.value) _
.ToArray
Dim anova As New AnovaTest()
anova.populate(observations, type:=AnovaTest.P_FIVE_PERCENT)
anova.findWithinGroupMeans()
anova.setSumOfSquaresOfGroups()
anova.setTotalSumOfSquares()
anova.divide_by_degrees_of_freedom()
Call base.cat("\n", env:=stream.ggplot.environment)
Call base.cat(anova.ToString, env:=stream.ggplot.environment)
Dim pvalue As Double = anova.singlePvalue
Dim tagStr As String = $"ANOVA, p-value={pvalue.ToString("G3")}"
Dim font As Font = CSSFont.TryParse(stream.theme.tagCSS).GDIObject(stream.g.Dpi)
Dim pos As New PointF(stream.scale.X.rangeMin + 10, stream.canvas.Padding.Top)
Call stream.g.DrawString(tagStr, font, Brushes.Black, pos)
End Sub可以看得见,ANOVA检验分析所使用的代码调用过程要比两两比对的t检验的代码要少很多了。在sciBASIC框架之中,进行ANOVA检验会需要用到AnovaTest
相应的,在ggplot程序包之中进行ANOVA检验计算的函数调用也是保持和R语言之中的ggplot2
# Add global annova p-value
stat_compare_means(method = "anova", label.y = 1600)添加特定组别t检验显著性标记
在上面所进行的可视化一般是两两比对的组别通过程序产生。现在假若仅需要对某两组数据做比较的话,我们可以通过geom_signif
''' <summary>
''' ## Create significance layer
''' </summary>
''' <returns></returns>
<ExportAPI("geom_signif")>
Public Function geom_signif(comparisons As list, Optional test As String = "t.test") As ggplotLayer
Return New ggplotSignifLayer With {
.comparision = comparisons,
.method = test
}
End Function可以看得到,上面的函数同样也是创建了一个ggplot的绘制图层对象,但是可以通过一个comparisons参数来手动控制所需要进行比较的组别信息。

ggplot(myeloma, aes(x = "molecular_group", y = "DEPDC1"))
# Add horizontal line at base mean
+ geom_hline(yintercept = mean(myeloma$DEPDC1), linetype="dash", line.width = 6, color = "red")
+ geom_boxplot(width = 0.65)
+ geom_jitter(width = 0.3)
+ ggtitle("DEPDC1 ~ molecular_group")
+ ylab("DEPDC1")
+ xlab("")
+ scale_y_continuous(labels = "F0")
+ stat_compare_means(method = "anova", label.y = 1600) # Add global annova p-value
+ geom_signif(list(vs = ["MAF","Proliferation"]))
+ theme(axis.text.x = element_text(angle = 45), plot.title = element_text(family = "Cambria Math", size = 16))
;从脚本的输出结果来看,程序仅针对了MAF和Proliferation这两个分组进行的差异显著性统计。
- R#环境中的热图颜色 - 2025年6月11日
- HE染色结果怎么看 - 2025年6月10日
- 脂质组学概述 - 2025年6月10日


No responses yet