估计阅读时长: 31 分钟Like the original R language it does, the R# system just provides a runtime to running […]
估计阅读时长: 69 分钟Read on CodeProject: https://www.codeproject.com/Articles/5338916/Introducing-Rsharp-language With many years of do scientific computing works by VB.NET language, I'm […]
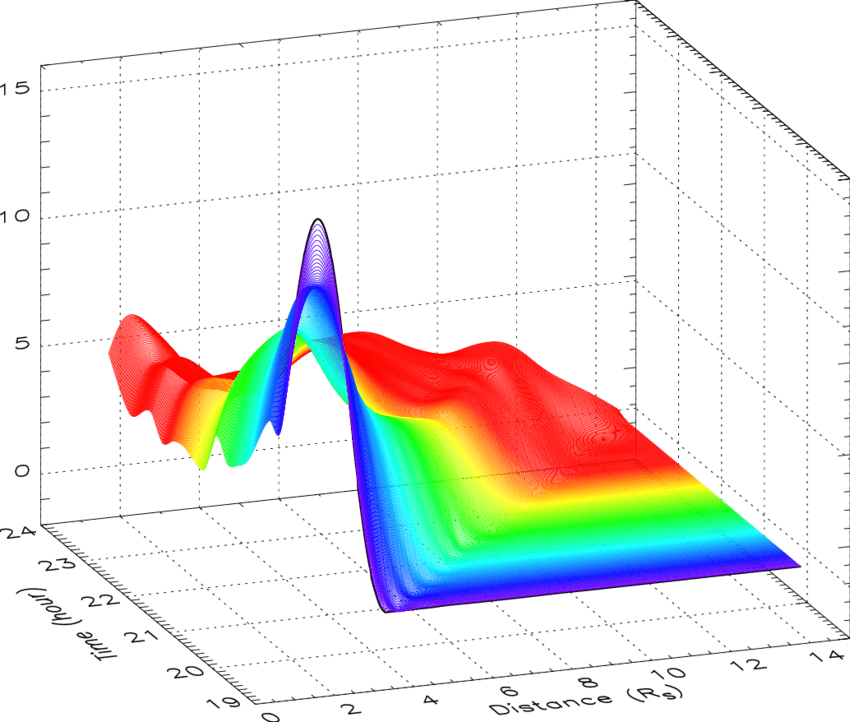
估计阅读时长: 7 分钟热图(Heat Map)是在二维空间中以颜色的形式显示一个现象的绝对量一种数据可视化技术。颜色的变化可能是通过色调或强度,给读者提供明显的视觉提示,说明现象是如何在空间上聚集或变化的。热图有两种完全不同的类别:聚集热图和空间热图。 在聚集热图中,幅度被排列成一个固定单元格大小的矩阵,其行和列是离散的现象和类别,行和列的排序是有意的,而且有些随意,目的是暗示聚集或描绘出通过统计分析发现的聚集。单元格的大小是任意的,但足够大,可以清晰可见。 相比之下,空间热图中某一量级的位置是由该量级在该空间中的位置所决定的,没有单元的概念,现象被认为是连续变化的。 Order by Date Name Attachments 2D-cubic-spline-interpolation-of-mass-profiles-from-1939-to-2354-UT-and-between-16 • 112 kB • 826 click […]
估计阅读时长: 7 分钟一般而言,进行全基因组的转录表达调控网络的建立,我们需要基于两个数据结果来完成: 目标基因的转录调控位点信息(Motif搜索结果,构成网络之中的节点) 转录调控位点相应的转录调控因子(Motif位点相关的转录调控因子,构成网络之中的边连接) Order by Date Name Attachments Xor • 271 kB • 684 click 2022年6月11日An […]
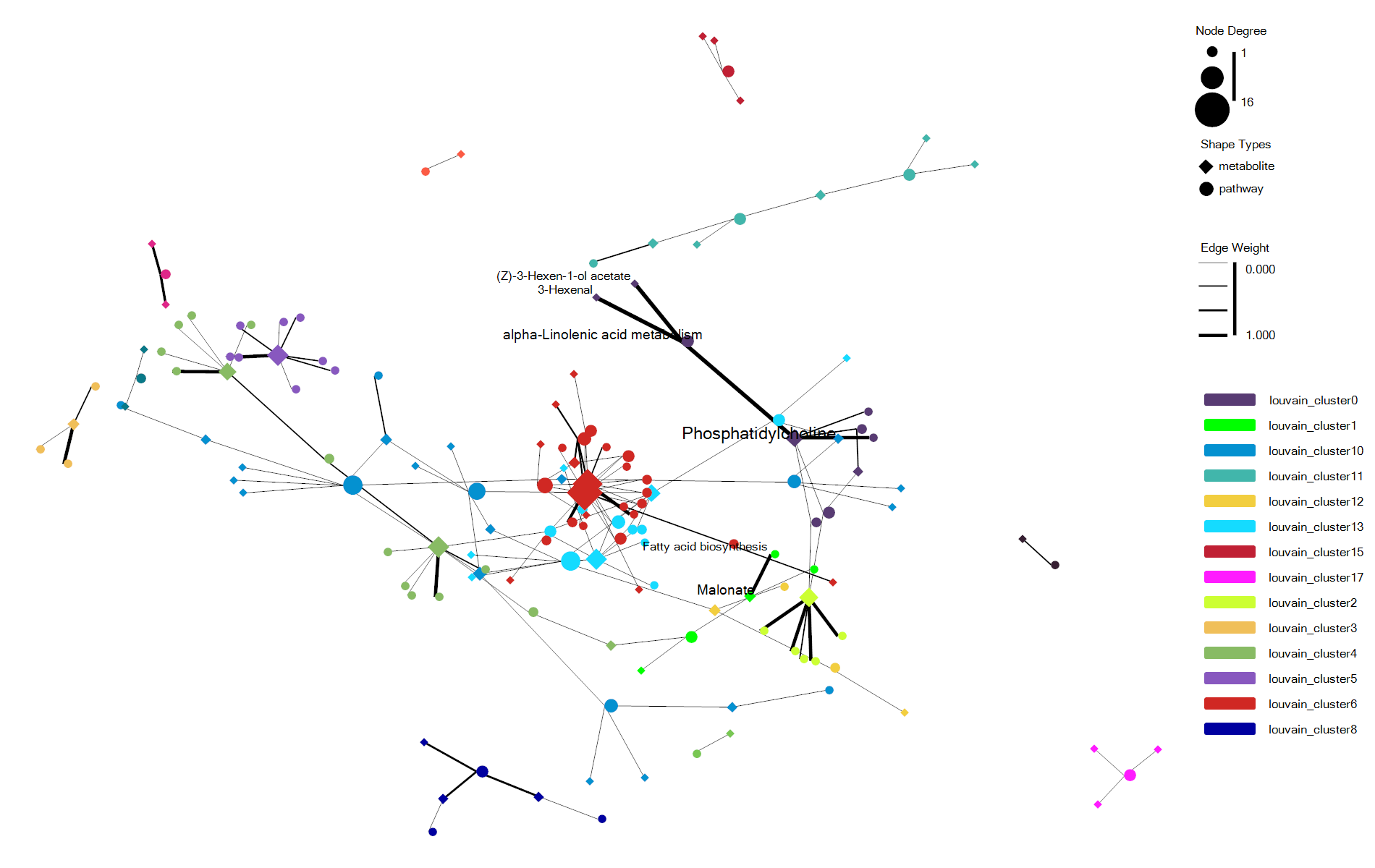
估计阅读时长: 7 分钟https://github.com/rsharp-lang/ggplot 在进行复杂关系的数据集进行可视化的时候,通过网络图的方式进行数据可视化可以让我们非常直观的借助于网络节点的聚集程度之类的布局信息了解到我们的复杂数据的关系结构信息。最近将R#语言之中的ggplot包进行网络可视化的代码库进行了一些更新。基于此功能更新工作,目前在ggplot程序包之中成功集成了ggraph程序包类似的网络可视化功能。在这里做了一些总结分享给大家。 Order by Date Name Attachments enrichNetwork_ggraph • 70 kB • 660 click 2022年6月1日enrichNetwork_ggraph2 • […]
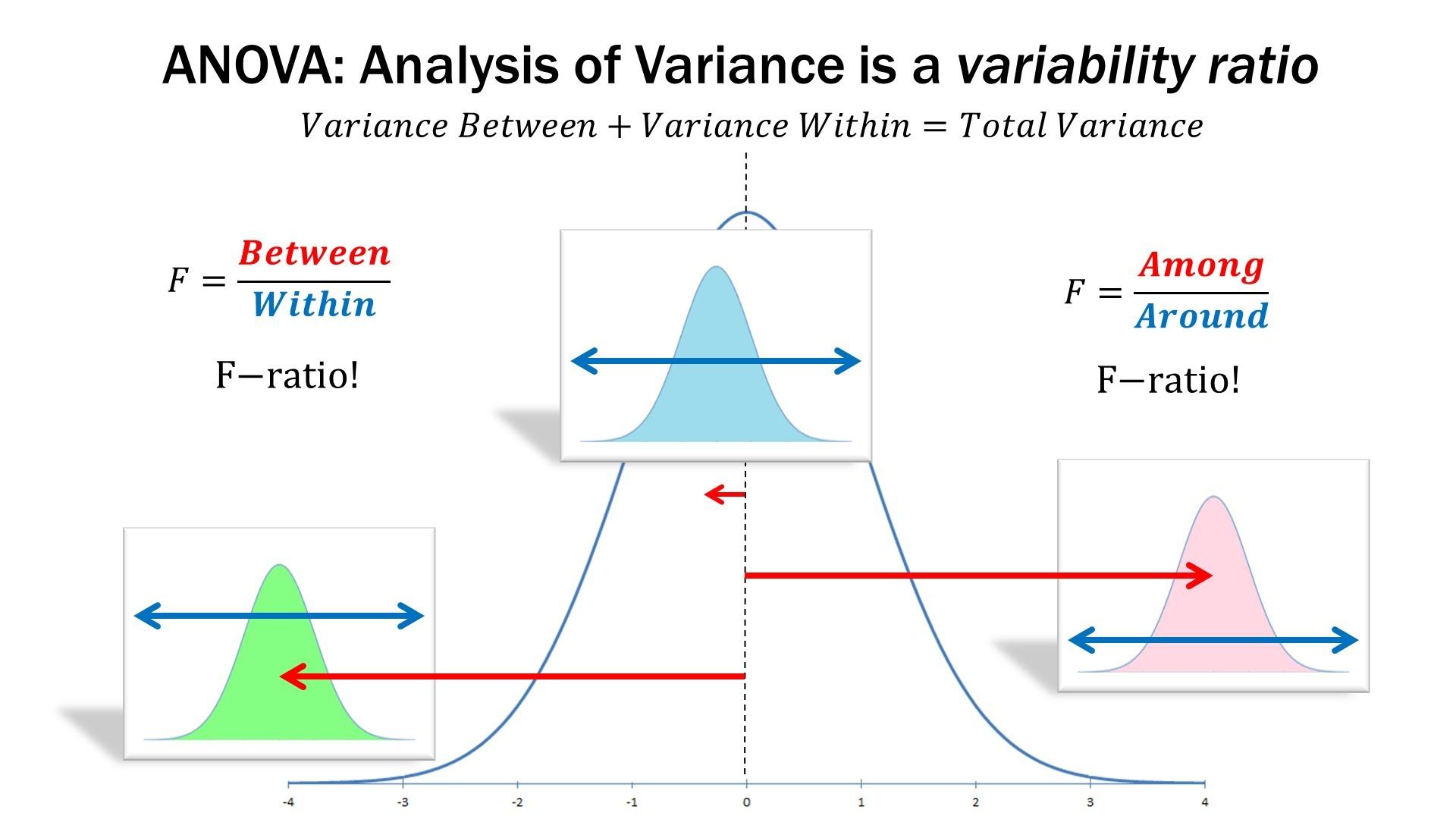
估计阅读时长: 14 分钟一般而言,如果我们在进行组学数据分析的时候,如果想要比较两组数据之间是否存在有差异性,一般是对两两比较的两组数据进行T-检验。但是在代谢组学数据分析领域内,则很多的组学数据分析情况为比较两组以上的数据,寻找差异的biomarker。那这个时候就需要使用上ANOVA统计检验方法了。 Order by Date Name Attachments anova • 105 kB • 849 click 2022年5月28日ANOVA-screen • 27 […]
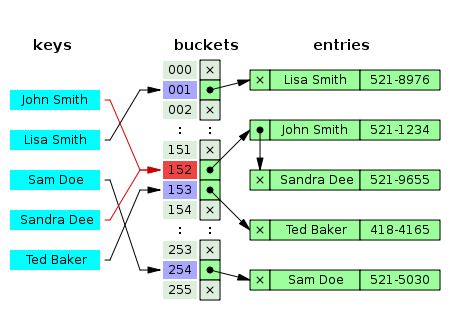
估计阅读时长: 8 分钟在之前的BioDeep代谢物数据库整合工作之中,所提取的代谢物注释信息的唯一编码是来自于数据库表之中的递增主键。由于数据库之中的递增主键的唯一编码值是与数据内容完全无关的数据,所以在基于图数据库做数据库整合的结果在两次整合操作之后,可能会因为先后输出顺序不一致的原因,得到的在关系型数据库中的唯一递增编号可能会完全不一样了。这个问题会对数据库更新操作造成非常大的困扰。 Order by Date Name Attachments 450px-Hash_table_5_0_1_1_1_1_1_LL • 26 kB • 637 click 2022年4月16日metadata-table • 58 […]
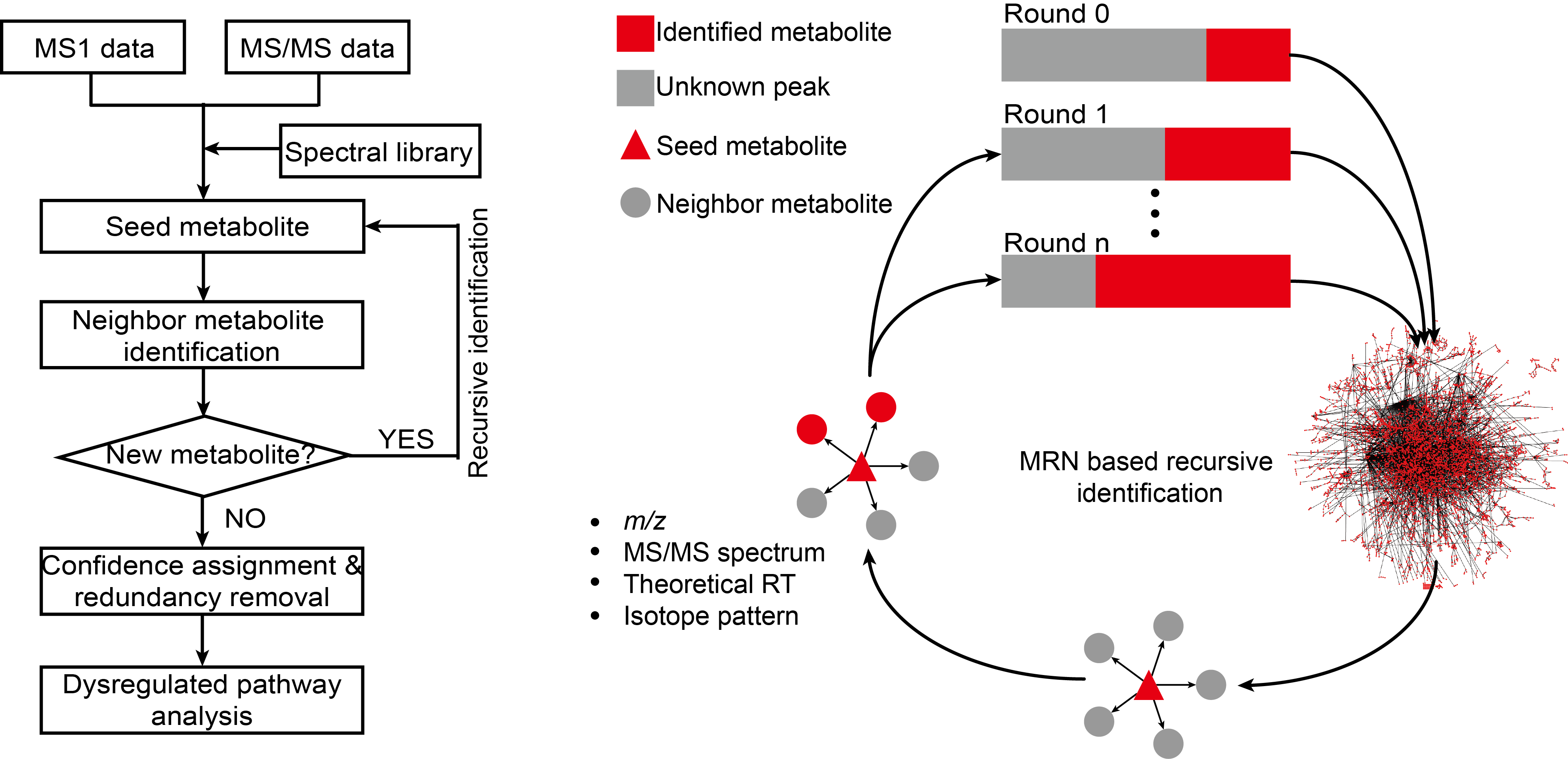
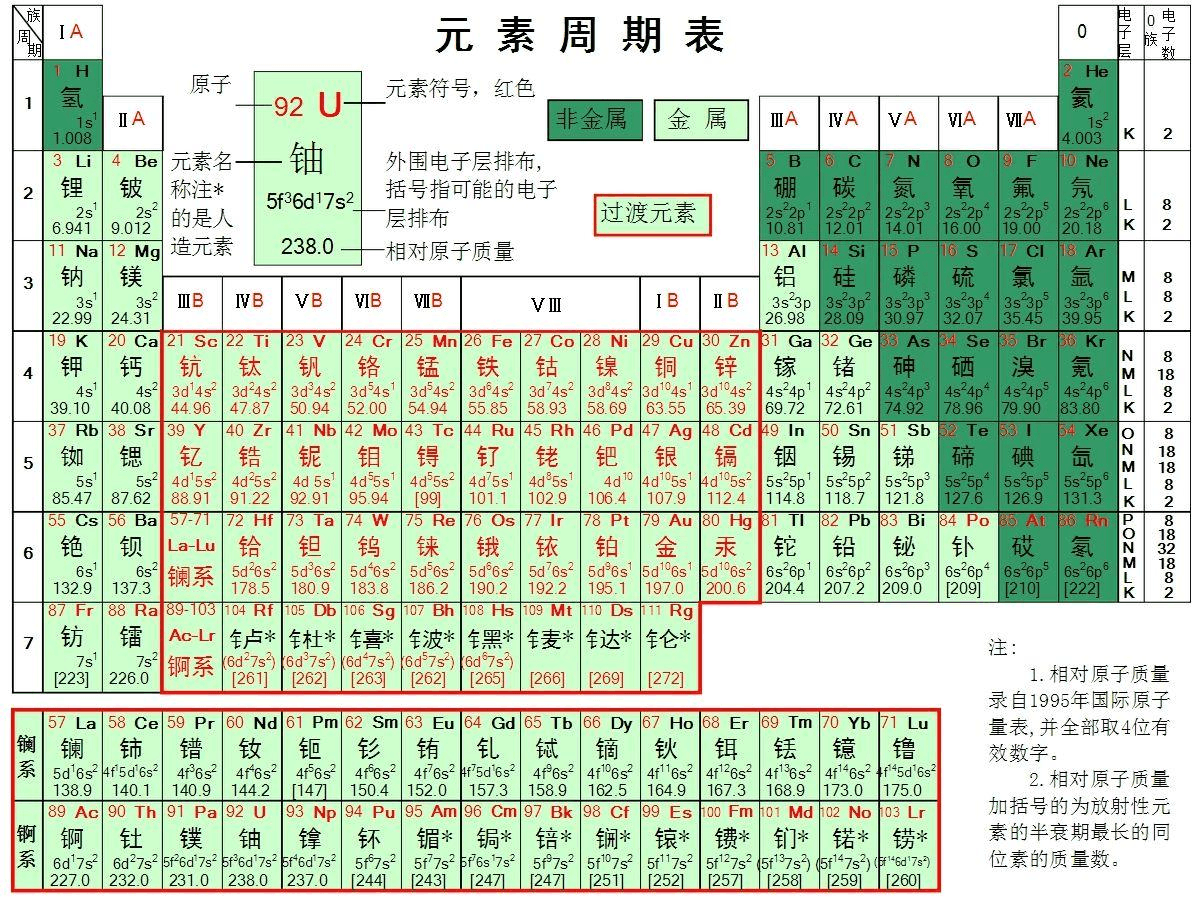
估计阅读时长: 5 分钟目前我们根据质谱数据进行代谢物ROI注释分析,很大一部分的工作是建立在已经可以被纯化的化合物的纯标准品所建立的标准品库数据的比对操作之上的。但是依赖于质谱参考谱图数据库所完成的代谢物注释分析,也仅能够得到很小的一部分结果,因为能够纯化或者合成的化合物在整个自然界中目前只占比较小的一部分。并且购买标准品也会需要耗费大量的实验室资金预算。 Order by Date Name Attachments The-Periodic-Table • 2 MB • 717 click 2022年3月20日Leucine[M+H]+ • 33 […]




[…] 对于基于ec number来生成层级数据,我们直接使用《酶EC编号结构解析》文章末尾所展示的层级数据生成函数来实现。 […]
[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]