估计阅读时长: 23 分钟Sequence Logo 是一种可视化 DNA 或蛋白质序列保守性的图形表示方法。每个位置(列)上的字母堆叠高度代表该位点的信息含量(以 bits 为单位),而每个字母的高度则与其在该位点出现的频率成正比。高信息量的位置字母堆得高,低信息量的位置则矮甚至接近零。Sequence Logo的绘制遵循信息熵原理,我们可以很直观的通过某一个位置的总高低来了解该处位置的信息含量有多少,高信息量的位置,字母堆的高,一般会出现某一个字符特别高,表明该处非常保守。 位置权重矩阵(Position Weight Matrix, PWM)是描述基因组调控因子结合位点序列模式的核心模型。它通过统计在结合位点序列中每个位置上各核苷酸(或氨基酸)出现的频率,来量化该位置对不同碱基的偏好程度。PWM通常以矩阵形式表示,行对应核苷酸(A、C、G、T/U),列对应序列中的位置,矩阵元素即为该位置该核苷酸相对于背景的权重得分。这一模型简洁且易于计算,因此在转录因子结合位点(TFBS)等调控元件的识别和表征中被广泛采用。 Order by Date Name […]
估计阅读时长: 7 分钟热图(Heat Map)是在二维空间中以颜色的形式显示一个现象的绝对量一种数据可视化技术。颜色的变化可能是通过色调或强度,给读者提供明显的视觉提示,说明现象是如何在空间上聚集或变化的。热图有两种完全不同的类别:聚集热图和空间热图。 在聚集热图中,幅度被排列成一个固定单元格大小的矩阵,其行和列是离散的现象和类别,行和列的排序是有意的,而且有些随意,目的是暗示聚集或描绘出通过统计分析发现的聚集。单元格的大小是任意的,但足够大,可以清晰可见。 相比之下,空间热图中某一量级的位置是由该量级在该空间中的位置所决定的,没有单元的概念,现象被认为是连续变化的。 Order by Date Name Attachments 2D-cubic-spline-interpolation-of-mass-profiles-from-1939-to-2354-UT-and-between-16 • 112 kB • 817 click […]
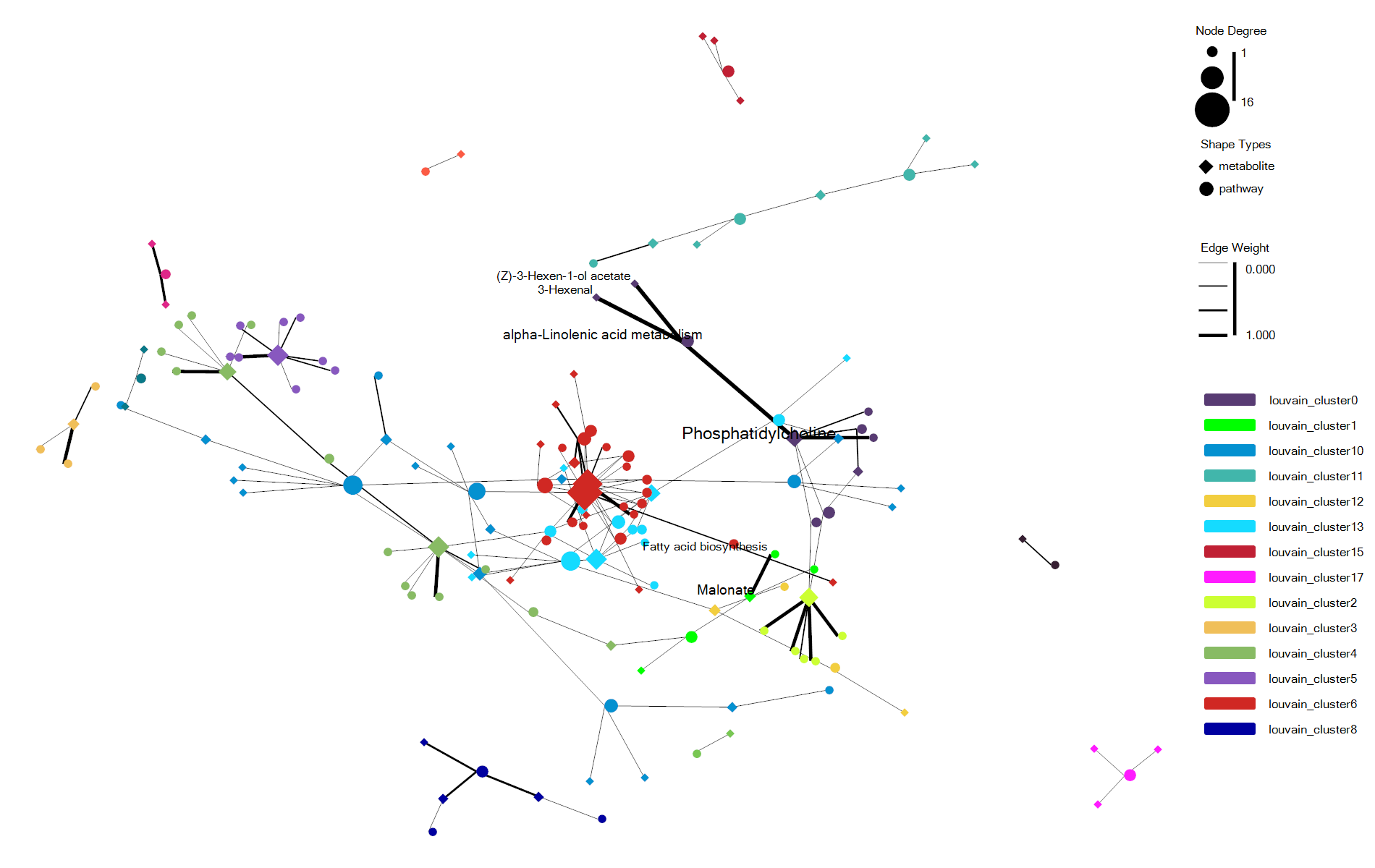
估计阅读时长: 7 分钟https://github.com/rsharp-lang/ggplot 在进行复杂关系的数据集进行可视化的时候,通过网络图的方式进行数据可视化可以让我们非常直观的借助于网络节点的聚集程度之类的布局信息了解到我们的复杂数据的关系结构信息。最近将R#语言之中的ggplot包进行网络可视化的代码库进行了一些更新。基于此功能更新工作,目前在ggplot程序包之中成功集成了ggraph程序包类似的网络可视化功能。在这里做了一些总结分享给大家。 Order by Date Name Attachments enrichNetwork_ggraph • 70 kB • 650 click 2022年6月1日enrichNetwork_ggraph2 • […]
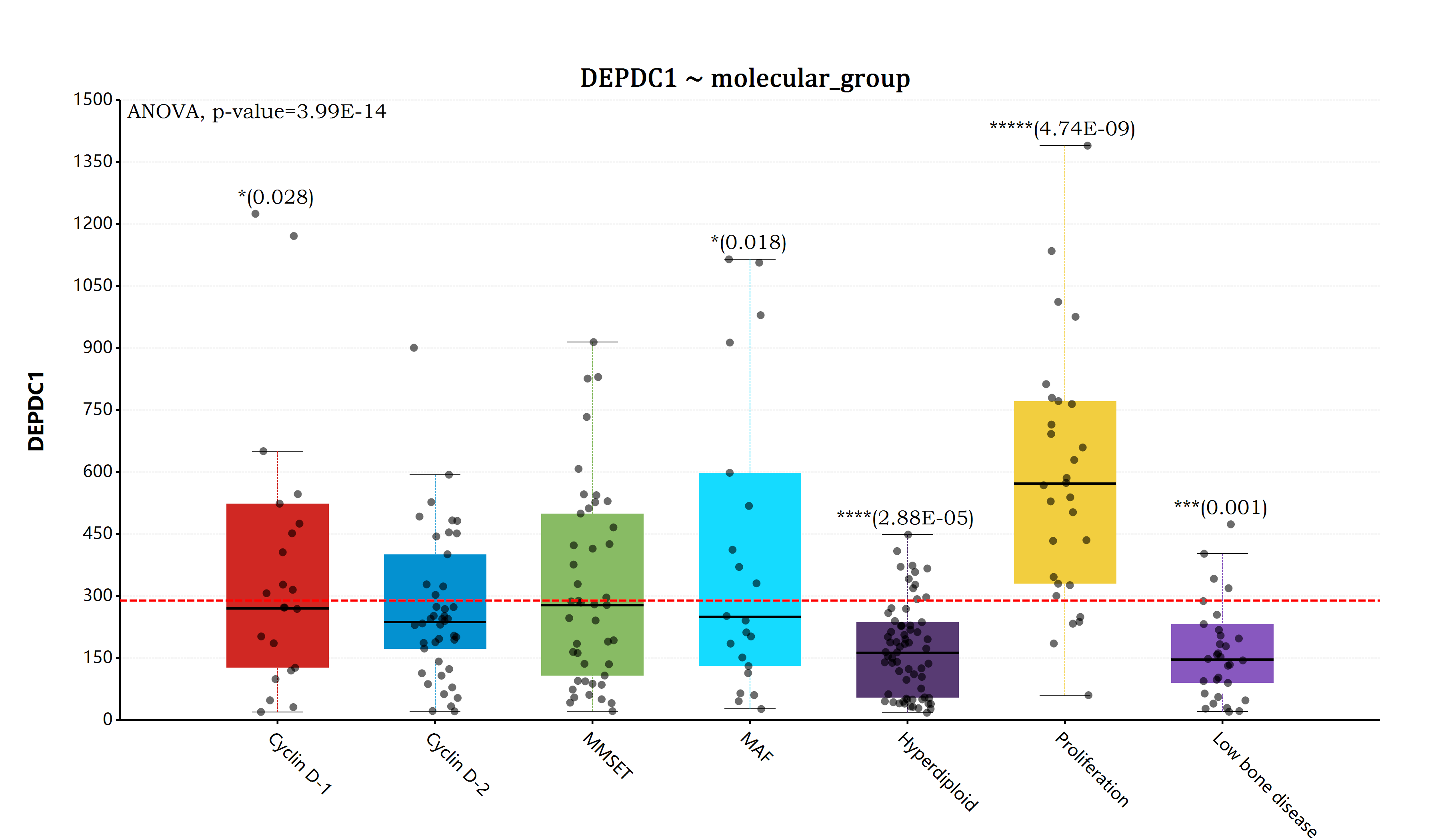
估计阅读时长: 14 分钟https://github.com/rsharp-lang/ggplot 在完成了前面所提到的ANOVA检验模块的代码开发编写工作之后,之前一直悬在我心里面的完善R#语言的ggplot统计作图功能的愿望现在终于实现了。在R#语言之中通过使用ggplot代码库进行相应的数据统计分析作图,目前已经变得和R语言之中的ggplot2程序包那样同样的简单和漂亮。 Order by Date Name Attachments myeloma_bar • 196 kB • 736 click 2022年5月29日myeloma_box • […]
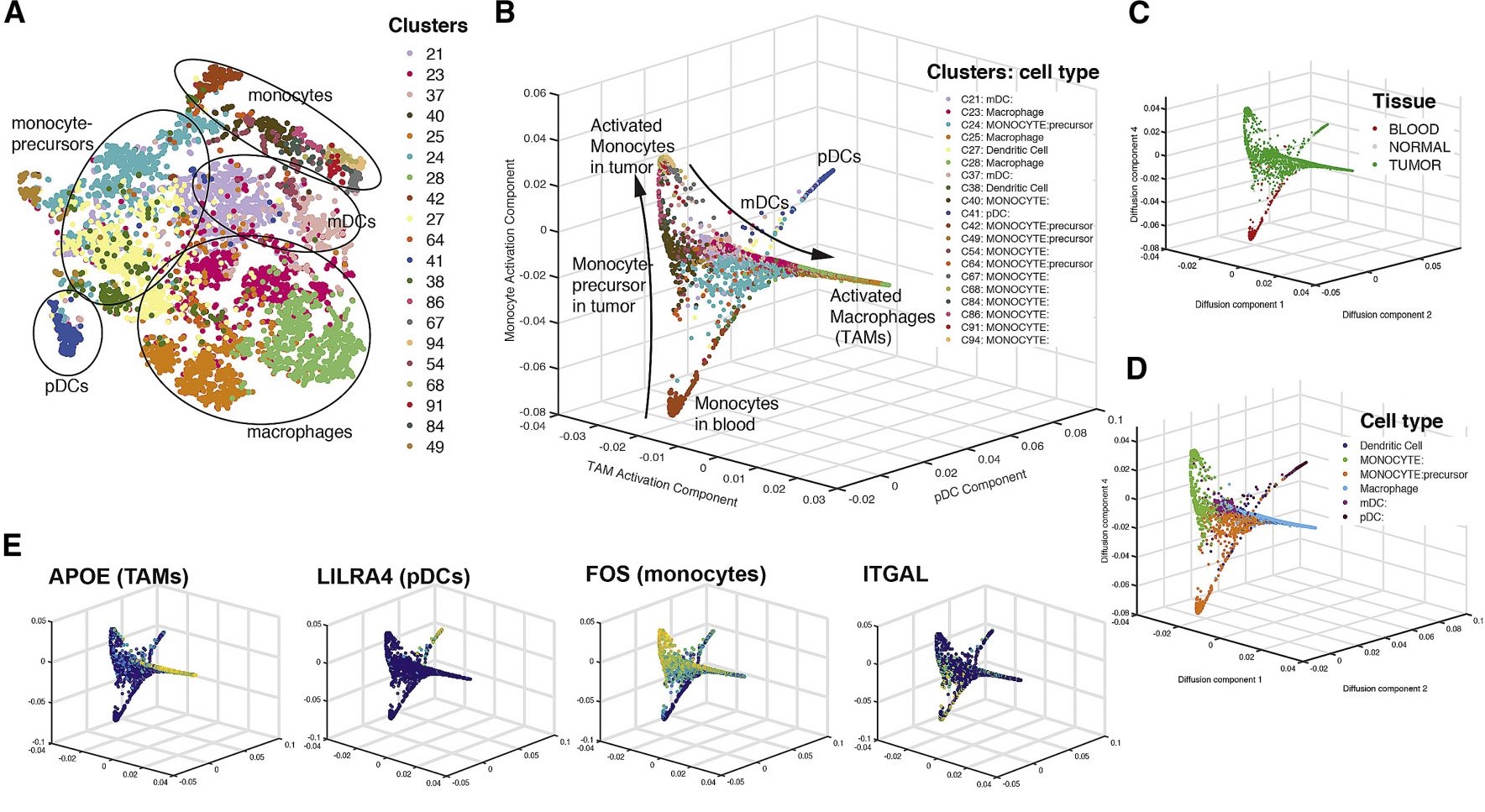
估计阅读时长: 14 分钟https://github.com/rsharp-lang/ggplot 之前在阅读一篇单细胞组学数据分析的文献,觉得在文献之中有一些三维散点图用于展示降维聚类结果的效果非常的好看。于是自己在R#语言之中的ggplot程序包的2D绘图的功能基础之上,进行了三维图形数据可视化功能的开发。 (A) t-SNE map projecting myeloid cells from BC1-8 patients (all tissues). Cells are colored […]
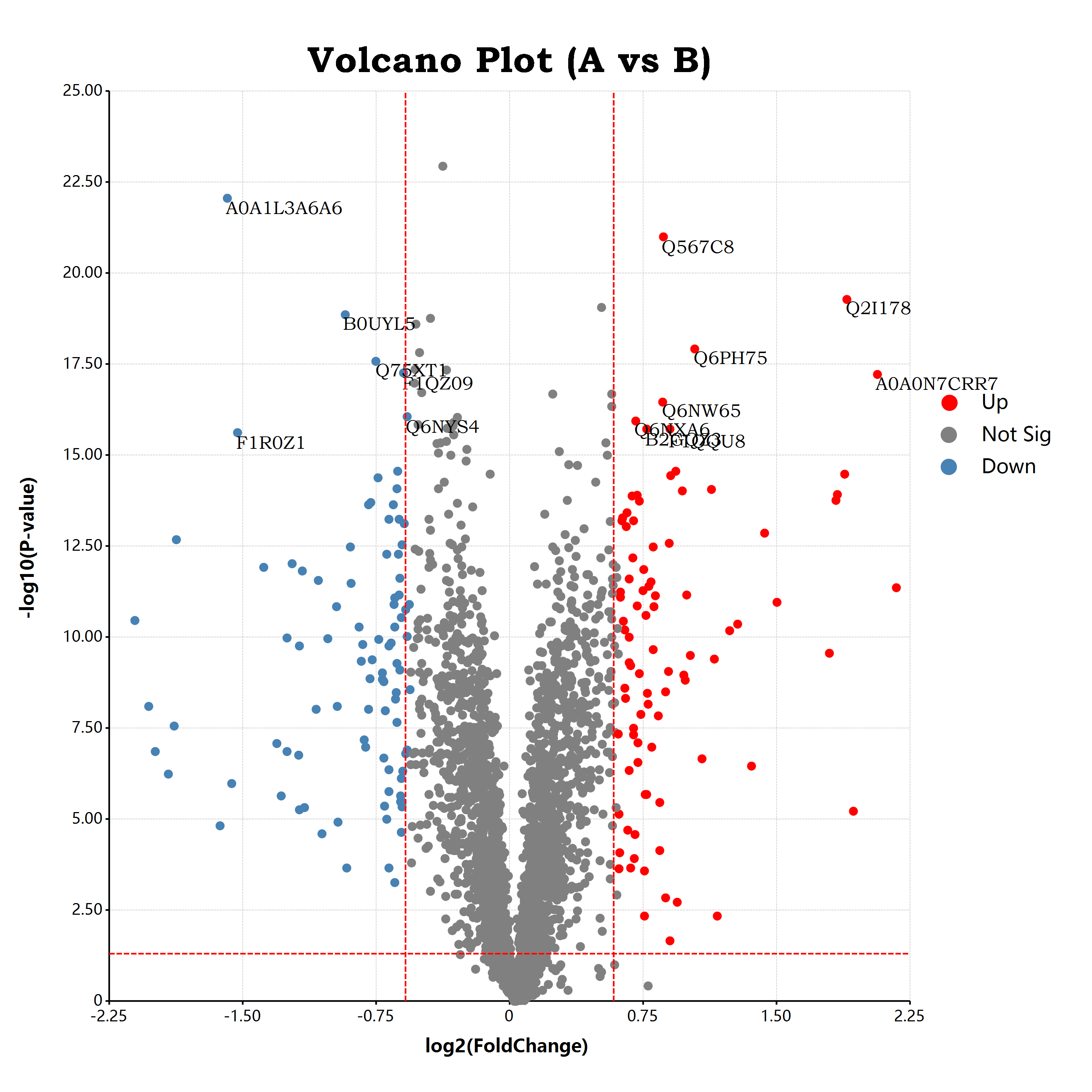
估计阅读时长: 17 分钟https://github.com/rsharp-lang/ggplot 接上一篇博客文章中谈到,我们已经通过R#语言之中的ggplot程序包绘制出了一个可以使用的火山图。在这里,我们将会通过在火山图上添加更多的可视化元素来为大家介绍R#语言之中的ggplot程序包的进阶使用方式。 Order by Date Name Attachments volcano • 651 kB • 729 click 2021年10月9日volcano • […]
估计阅读时长: 11 分钟https://github.com/rsharp-lang/ggplot 在生物信息学中的组学数据分析领域内,有一个非常常见的数据可视化图表:应用于可视化两两组别比对结果的火山图。在火山图之中,X坐标轴一般是log2FC,纵坐标Y轴,则一般是t检验的pvalue的-log10转换之后的值。由于fold change有大于1的值,A/B大于1,表示A的表达量高于B的表达量,反之小于一表示A的表达量低于B的表达量。这样子fold change经过log2转换之后,就会出现负数,散点一般呈轴对称分布在X=0的位置周围。这样子绘制出来的散点图就有点类似于火山喷发的样子了。 Order by Date Name Attachments a679af1eb9ffbfbad48c18d563ea51f3 • 45 kB • 701 click […]


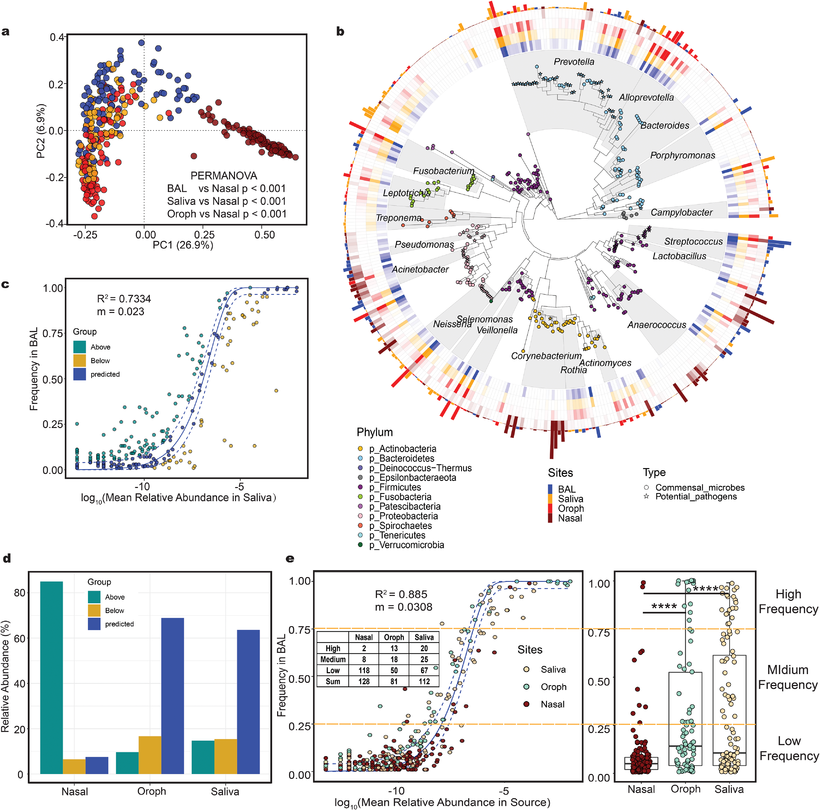

[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]