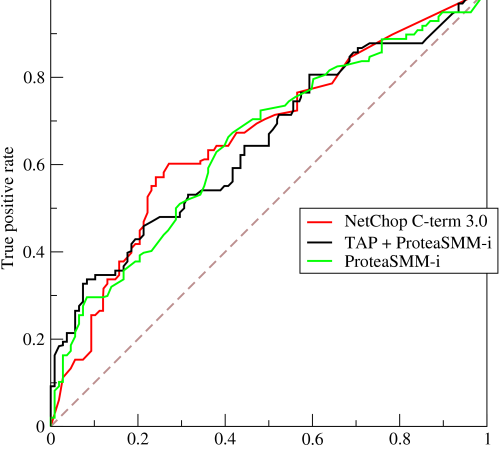
估计阅读时长: 8 分钟https://github.com/rsharp-lang/R-sharp 对于0,1两类分类问题,一些分类器得到的结果往往不是0,1这样的标签。如神经网络得到诸如0.5,0.8这样的分类结果。这时,我们人为取一个阈值,比如0.4,那么小于0.4的归为0类,大于等于0.4的归为1类,可以得到一个分类结果。同样,这个阈值我们可以取0.1或0.2等等。 Order by Date Name Attachments ROC • 221 kB • 752 click 2021年6月28日Roccurves • […]
估计阅读时长: 23 分钟https://github.com/rsharp-lang/R-sharp 降维是将数据由高维约减到低维的过程而用来揭示数据的本质低维结构。它作为克服“维数灾难”的途径在这些相关领域中扮演着重要的角色。在过去的几十年里,有大量的降维方法被不断地提出并被深入研究,其中常用的包括传统的降维算法如PCA和MDS;流形学习算法如UMAP、t-SNE、ISOMAP、LE以及LTSA等。 Order by Date Name Attachments MNIST-LabelledVectorArray-60000x100 • 230 kB • 849 click 2021年6月27日MNIST-LabelledVectorArray-60000x100Euclidean_Distance • […]
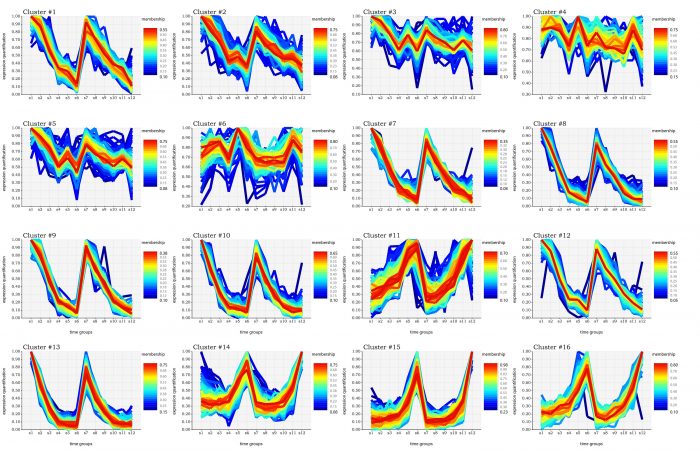
估计阅读时长: 11 分钟https://github.com/SMRUCC/GCModeller 在R语言之中,存在着一个用于进行表达数据的时间序列分析的程序包:TCseq。TCseq的全称为Time course sequencing,即时间序列分析,通过对表达矩阵进行时间上的模糊CMeans聚类,得到表达变化趋势一致的基因列表,进行基因表达的时间趋势分析。 在GCModeller之中,我仿照着TCseq程序包,自己编写了一个时间序列的聚类与可视化分析的R#程序包模块,在这里介绍给大家。 Order by Date Name Attachments Gene expression pattern visualization • 2 […]

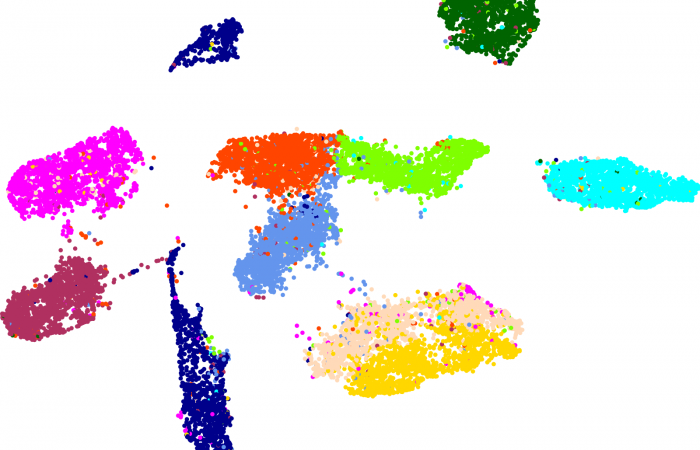

[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]