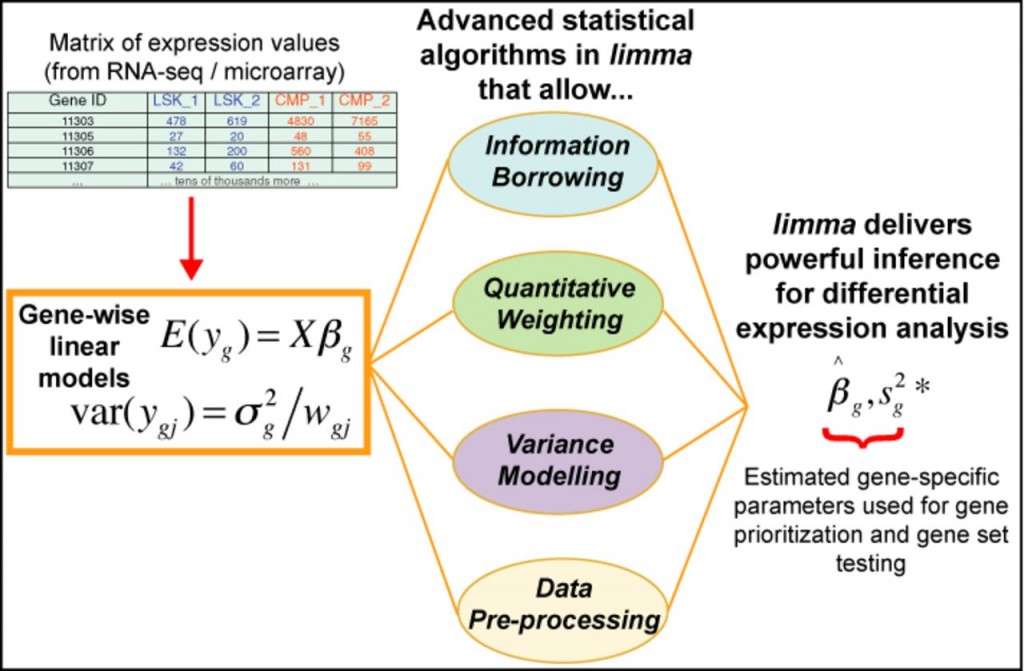
估计阅读时长: 22 分钟limma(Linear Models for Microarray Data)是一个基于R语言的Bioconductor包,最初用于微阵列数据的差异表达分析,现已扩展支持RNA-seq数据。其核心思想是利用线性模型(Linear Models)对基因表达数据进行建模,并结合经验贝叶斯(Empirical Bayes)方法在小样本情况下增强统计推断的稳健性。 Order by Date Name Attachments limma • 119 kB […]
估计阅读时长: 30 分钟零分布(null distribution)是指在假设零假设(null hypothesis)成立的情况下,某个统计量随机取值的概率分布。在统计假设检验中,我们通常提出一个零假设(例如“两组数据没有显著差异”或“观察到的模式仅由随机因素造成”),然后根据观测数据计算一个检验统计量。零分布描述了这个统计量在零假设为真时的分布情况。通过将实际观测到的统计量与零分布进行比较,我们可以计算出P-value:即在零假设下,出现等于或更极端观测结果的概率。如果P-value很小(例如低于预设的显著性水平α),我们就认为零假设不太可能成立,从而拒绝零假设,认为观测结果是统计显著的。 Order by Date Name Attachments image-2 • 66 kB • 206 click 2025年12月16日NULL-pvalue […]
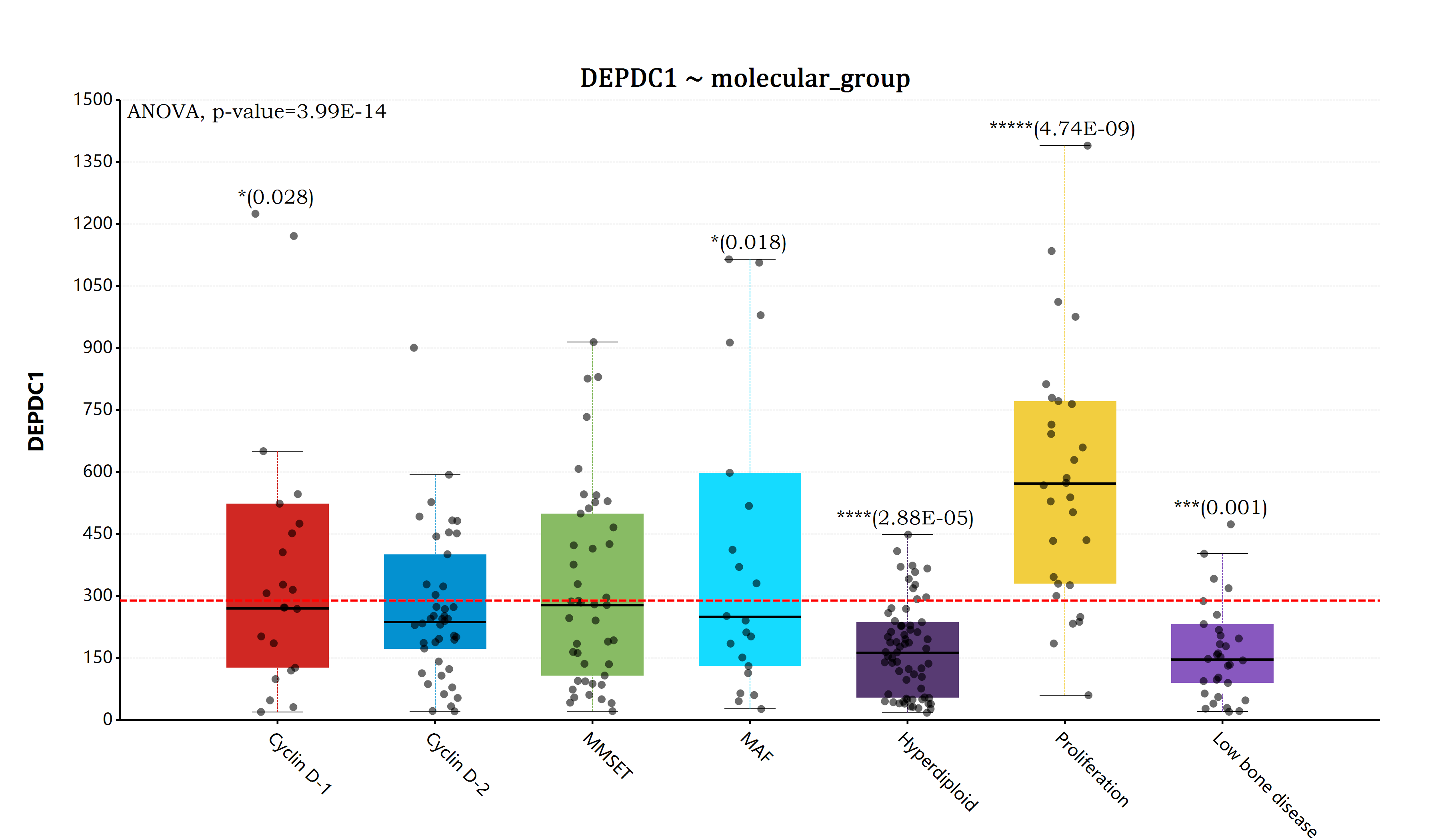
估计阅读时长: 14 分钟https://github.com/rsharp-lang/ggplot 在完成了前面所提到的ANOVA检验模块的代码开发编写工作之后,之前一直悬在我心里面的完善R#语言的ggplot统计作图功能的愿望现在终于实现了。在R#语言之中通过使用ggplot代码库进行相应的数据统计分析作图,目前已经变得和R语言之中的ggplot2程序包那样同样的简单和漂亮。 Order by Date Name Attachments myeloma_bar • 196 kB • 742 click 2022年5月29日myeloma_box • […]
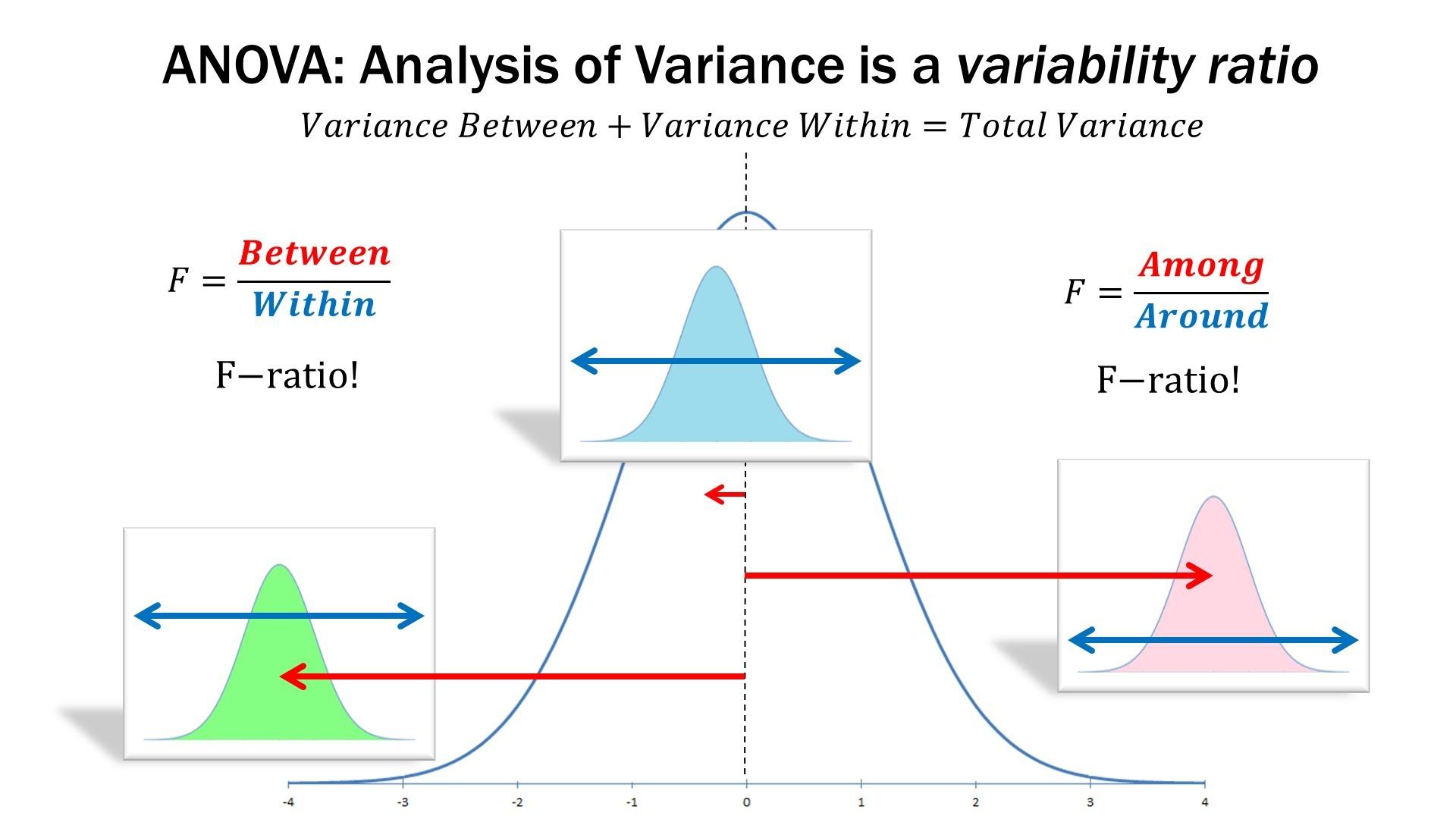
估计阅读时长: 14 分钟一般而言,如果我们在进行组学数据分析的时候,如果想要比较两组数据之间是否存在有差异性,一般是对两两比较的两组数据进行T-检验。但是在代谢组学数据分析领域内,则很多的组学数据分析情况为比较两组以上的数据,寻找差异的biomarker。那这个时候就需要使用上ANOVA统计检验方法了。 Order by Date Name Attachments anova • 105 kB • 844 click 2022年5月28日ANOVA-screen • 27 […]
估计阅读时长: 7 分钟F统计量是群体遗传学中由Sewall Wright提出的重要统计量,用于衡量遗传变异在群体中的分布情况。它提供了对群体遗传结构和遗传分化的定量描述。F统计量主要有三种类型:Fis、Fit和Fst,分别反映个体内的、总体的和群体间的遗传分化。F统计量在群体遗传学中通常指的是Fst(Fixation Index,固定指数),它是一个衡量群体间遗传差异的指标。Fst的值范围从0到1,其中0表示群体间没有差异,1表示群体间完全分离。在群体遗传学研究中,Fst常用于评估群体的遗传多样性、群体间的迁移率以及自然选择的压力等。 Order by Date Name Attachments 41598_2021_92984_Fig1_HTML • 2 MB • 717 click 2022年5月28日p1 […]
估计阅读时长: 2 分钟在BILIBILI上观看视频:《【GCModeller教程】基因组功能富集计算原理》 Order by Date Name Attachments 20190818_GSEA_release.mp4_20190921_225144.467 • 226 kB • 699 click 2021年5月30日Fisher Exact Test […]

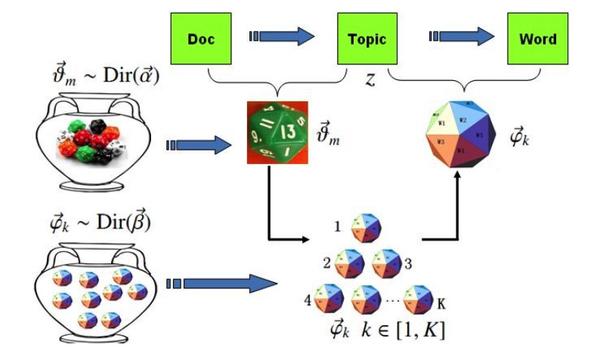
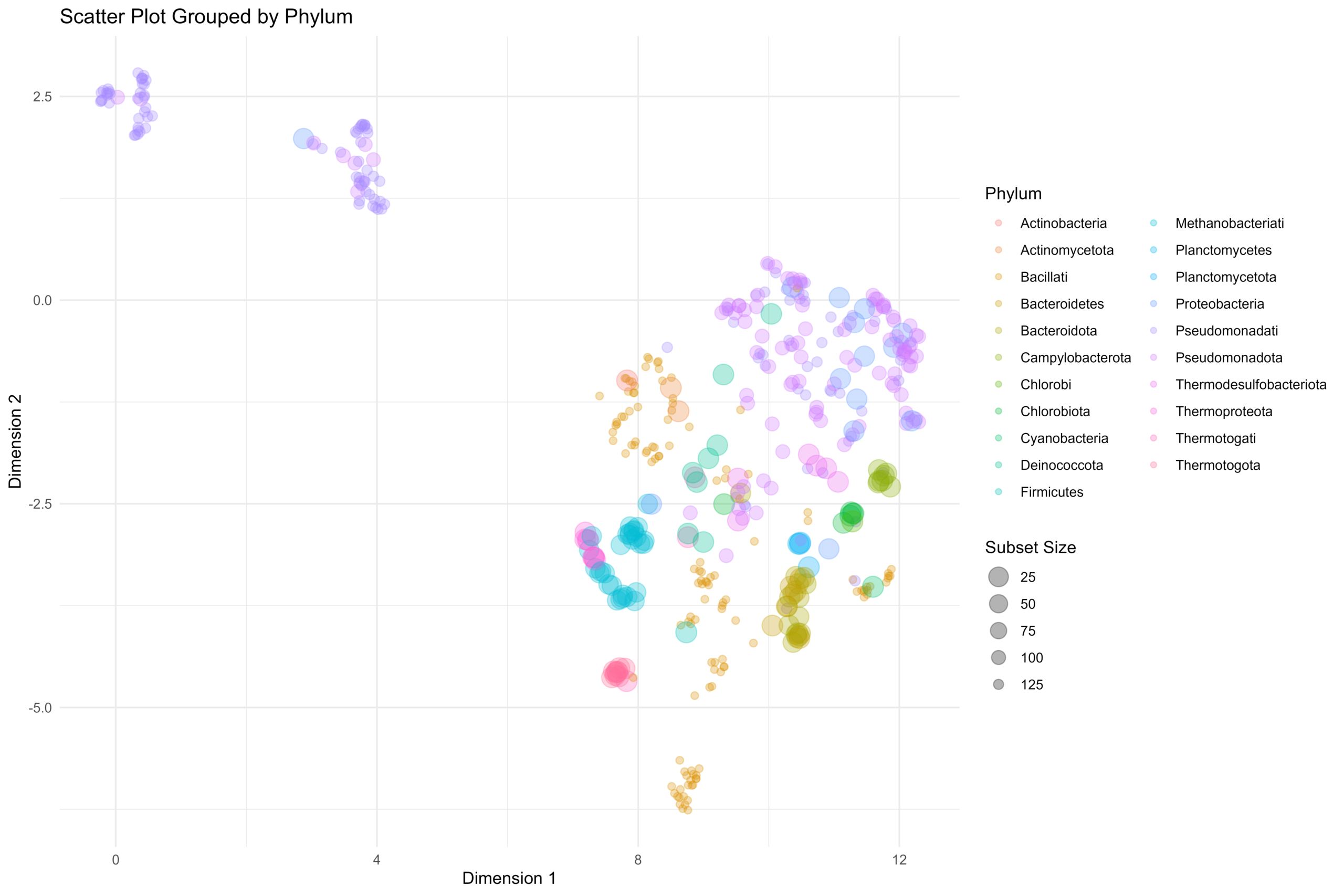



[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]