估计阅读时长: 4 分钟 在代谢组学领域内,LCMS原始数据分析一般分为非靶向全扫原始数据,以及仅针对某些离子进行扫描的MRM靶向质谱数据。虽然二者都是基于LCMS方法进行实验,但是MRM靶向数据由于在事先已经通过实验确定,得到了Q1和Q3离子对信息,所以可以仅针对某一些特定代谢物进行检测。因为MRM数据是针对于某些代谢物检测的靶向数据,所以其XIC谱图在没有同分异构体存在的情况下,一般是很纯净的目标化合物的检测结果数据。所以在原始数据分离,定量计算方面都要比非靶向全扫结果数据要容易很多。 Order by Date Name Attachments xcms-logo-white • 183 kB • 142 click 01.07.2022lcmspreproc_slides_1.2 • 136 […]
估计阅读时长: 2 分钟 https://mzkit.org/ 代谢组文章整个坎坷的发表经历中,大家可能都会遇到的一个老大难问题就是我们有时候会需要从原始数据中得到物质注释结果的二级质谱图数据。对于熟悉xcms程序包的同学,获取二级质谱图可能会比较容易:无非就是加载原始数据,然后按照m/z和rt找到对应的二级scan就好了。但是,这种方法会需要编写脚本来完成。 Order by Date Name Attachments download • 33 kB • 189 click 04.08.2021[278][MS_MS] FTMS […]
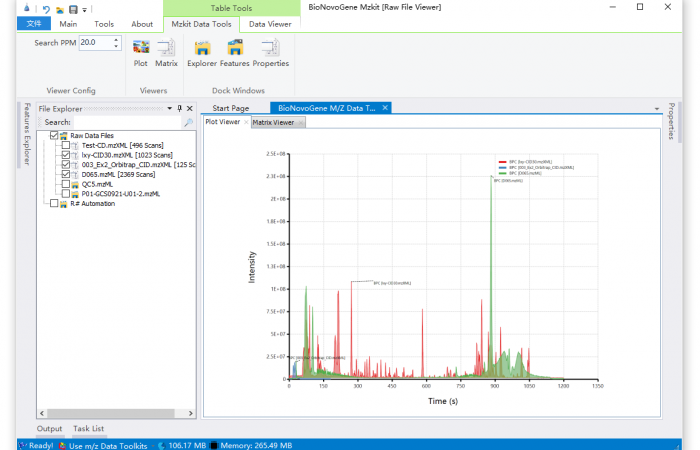
估计阅读时长: 3 分钟 https://mzkit.org mzkit软件是我最近开发的一款开源的代谢组学领域内的原始数据文件查看工具。开发mzkit软件的初衷是为了更方便的查看很大的非靶向原始数据文件:因为在开发mzkit软件之前,在开发LCMS的代谢物注释脚本或者建立标准品库数据的时候,如果我想要查看或者导出文件中的一些质谱图碎片信息,会需要通过R环境之中的xcms程序包编程来完成。通过R脚本来查看原始数据文件,非常的不方便。所以就有了mzkit软件项目的诞生。 Order by Date Name Attachments BPC_overlay • 114 kB • 180 click 01.07.2021LCMS_scanTree • […]
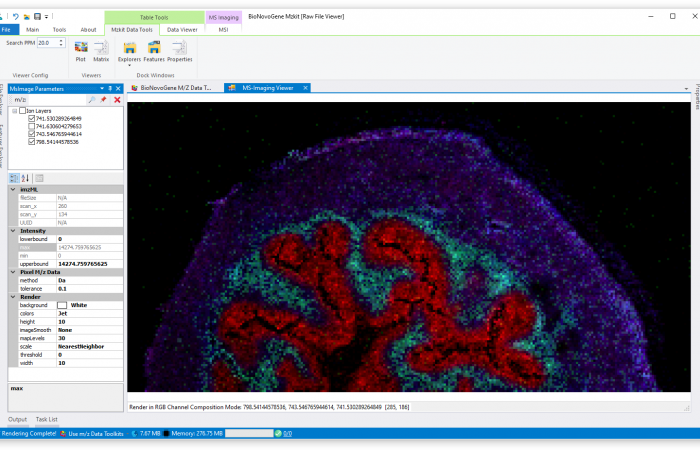
估计阅读时长: 3 分钟 http://mzkit.org/ 质谱成像是以质谱技术为基础的成像方法,该方法通过质谱直接扫描生物样品成像,可以在同一张组织切片或组织芯片上同时分析数百种分子的空间分布特征。 Order by Date Name Attachments HR2MSI mouse urinary bladder S096 - optical image • […]


Hi, I log on to your new stuff like every week. Your humoristic style is witty, keep it up
waiting and waiting.
Cool + for the post
老师提到的这个问题,我这几天会写一个使用教程,老师后面可以用教程中对应的demo示例来了解下功能的使用
空间Spot结果在下载到的mzkit文件夹中有示例吗?我试了试,不是10X结果中的tissue_positions_list.csv(软件选择此文件,直接error);在默认结果中也没找到类似的文件;