估计阅读时长: 8 分钟为了能够稳定的使用Ubuntu系统作为我的测试环境,我专门安装了Ubuntu的2404 LTS版本。但是发现虽然是LTS版本,仍然会因为系统更新而出现各种莫名奇妙的BUG。例如,在今天我为了安装软件,使用apt upgrade更新系统环境之后,出现了下面的消息: Order by Date Name Attachments ubuntu-24-04-noble-numbat • 66 kB • 443 click 2025年6月7日ti8S1 […]
估计阅读时长: 2 分钟Docker镜像信息 GCModeller以R#语言的软件包的形式提供给客户使用,相应的R#语言的分析环境以Docker镜像的形式进行打包盒发布,Docker的基础镜像为ubuntu 22.04。 dotnet环境:.NET 6 R#语言安装位置:/usr/local/bin R#程序包安装列表: 索引 包名称 Github 1 GCModeller https://github.com/SMRUCC/GCModeller 2 REnv https://github.com/rsharp-lang/R-sharp […]
估计阅读时长: 6 分钟CentOS查看系统版本信息 cat /etc/redhat-release # CentOS Stream release 8 cat /proc/version # Linux version 4.18.0-489.el8.x86_64 (mockbuild@x86-05.stream.rdu2.redhat.com) (gcc […]
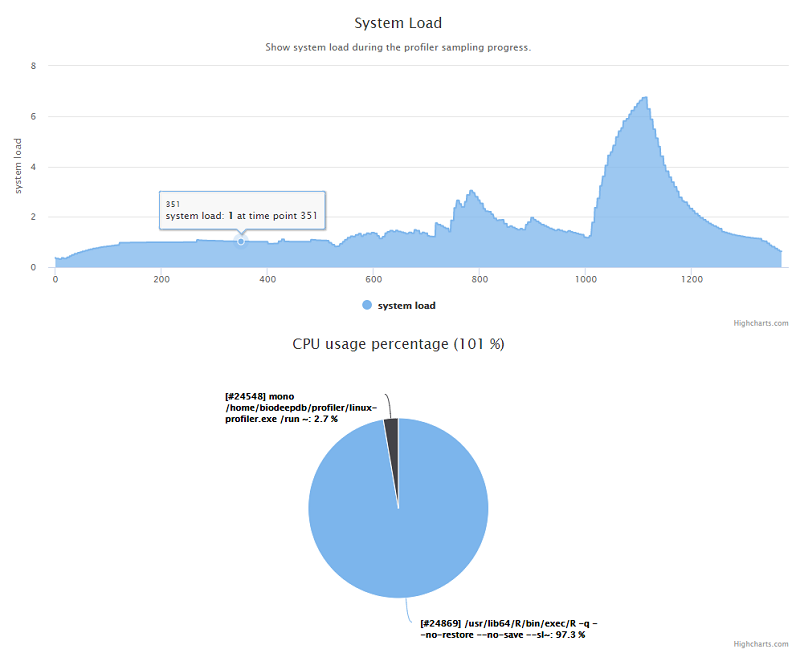
估计阅读时长: 6 分钟https://github.com/xieguigang/linux-profiler 废话不多说,首先给出一个 demo报告链接 给大家看看这个小工具的成品输出。 在去年的工作中,因为公司需要购买新的服务器做集群计算,需要一个工具来记录之前的服务器在数据分析上的性能瓶颈。于是花了两天的时间赶出来了这个专门应用于Linux系统的性能记录工具。这个小工具是一个开源项目,大家可以在Github上阅读这个开源项目(linux-profiler)的源代码。 Order by Date Name Attachments systemLoad • 53 kB • 705 […]





[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]