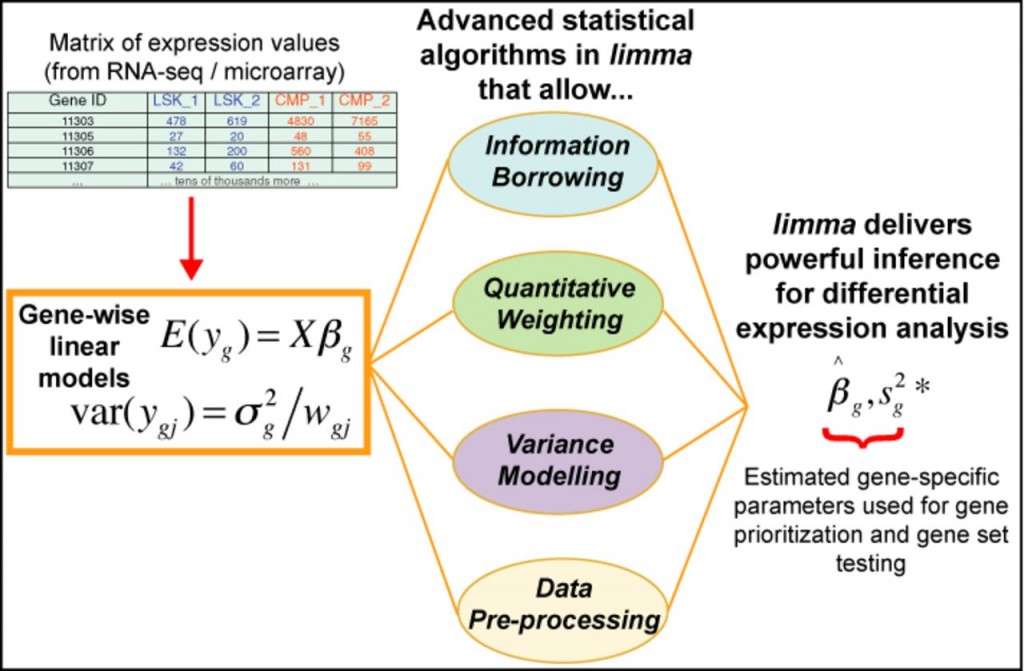
估计阅读时长: 22 分钟limma(Linear Models for Microarray Data)是一个基于R语言的Bioconductor包,最初用于微阵列数据的差异表达分析,现已扩展支持RNA-seq数据。其核心思想是利用线性模型(Linear Models)对基因表达数据进行建模,并结合经验贝叶斯(Empirical Bayes)方法在小样本情况下增强统计推断的稳健性。 Order by Date Name Attachments limma • 119 kB […]
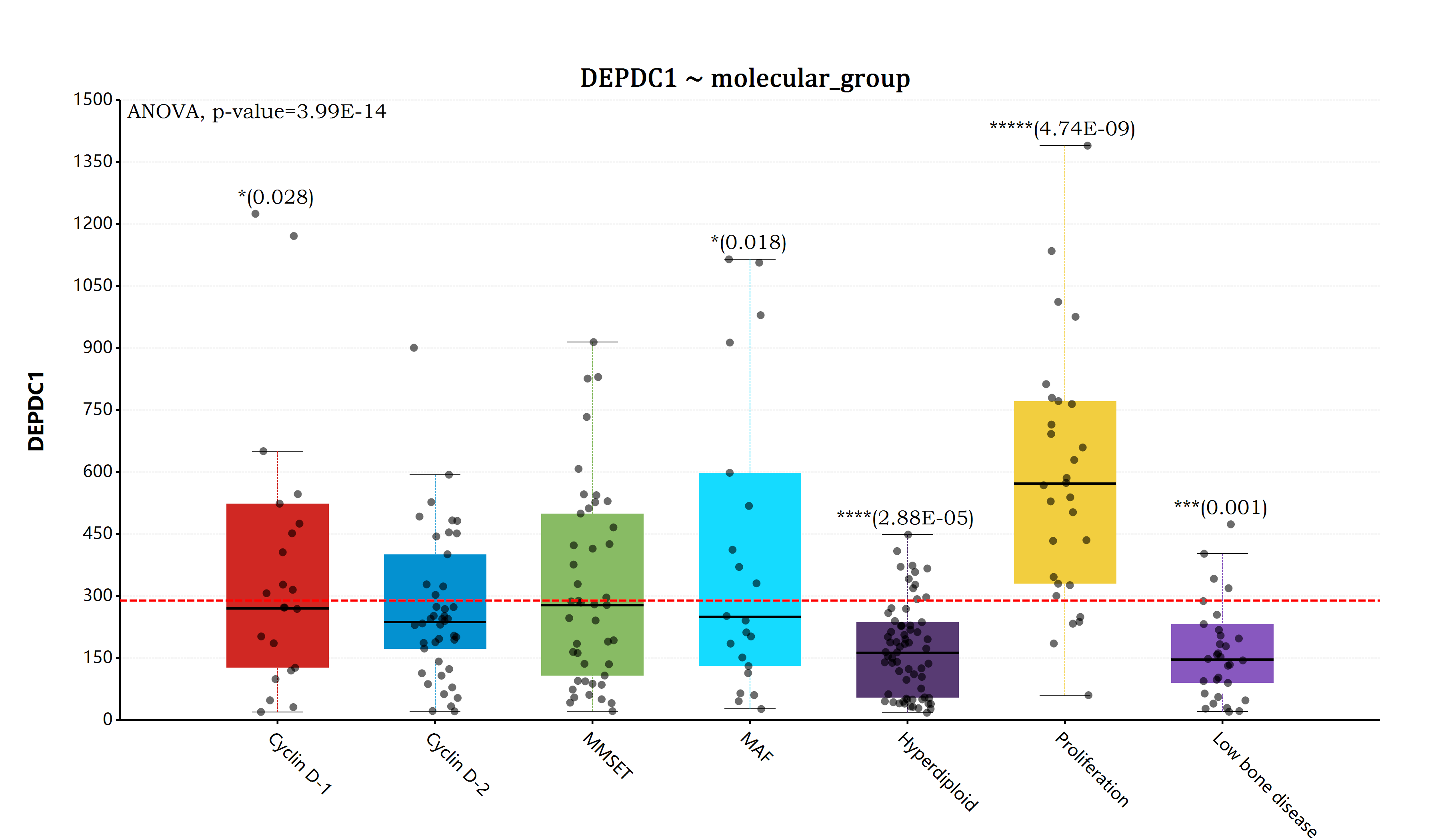
估计阅读时长: 14 分钟https://github.com/rsharp-lang/ggplot 在完成了前面所提到的ANOVA检验模块的代码开发编写工作之后,之前一直悬在我心里面的完善R#语言的ggplot统计作图功能的愿望现在终于实现了。在R#语言之中通过使用ggplot代码库进行相应的数据统计分析作图,目前已经变得和R语言之中的ggplot2程序包那样同样的简单和漂亮。 Order by Date Name Attachments myeloma_bar • 196 kB • 758 click 2022年5月29日myeloma_box • […]


[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 对于基于ec number来生成层级数据,我们直接使用《酶EC编号结构解析》文章末尾所展示的层级数据生成函数来实现。 […]
[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?