估计阅读时长: 11 分钟在进行热图的渲染的时候,我们需要首先将需要进行渲染的数据转换为一个0到1之间的灰度值,然后基于所设定的颜色列表,将灰度值映射为颜色列表的索引号,获取某一个灰度对应的颜色,从而完成对热图的渲染过程。在这个过程中,假若我们是针对热图需要获取得到一个连续的颜色列表,则我们还需要使用插值算法针对基础的关键颜色列表进行插值计算,生成调色板。 Order by Date Name Attachments volcano_ggthemes_Traffic • 17 kB • 450 click 2025年6月12日volcano_ggthemes_excel_Ion_Boardroom • 15 […]
估计阅读时长: 11 分钟给定一组n个字符串数组,找到包含给定集合中每个字符串的最小字符串作为子字符串。我们可以假设这个字符串数组中没有字符串是另一个字符串的子字符串。那么基于上面的描述,我们就可以得到下面所示的问题求解目标: let arr[] = ["catg", "ctaagt", "gcta", "ttca", "atgcatc"] // output: gctaagttcatgcatc 上面的问题描述实际上是一个最短超字符串问题(shortest common superstring) Order […]

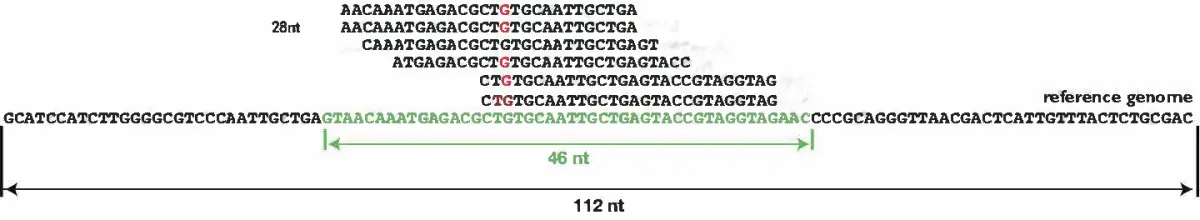

[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]