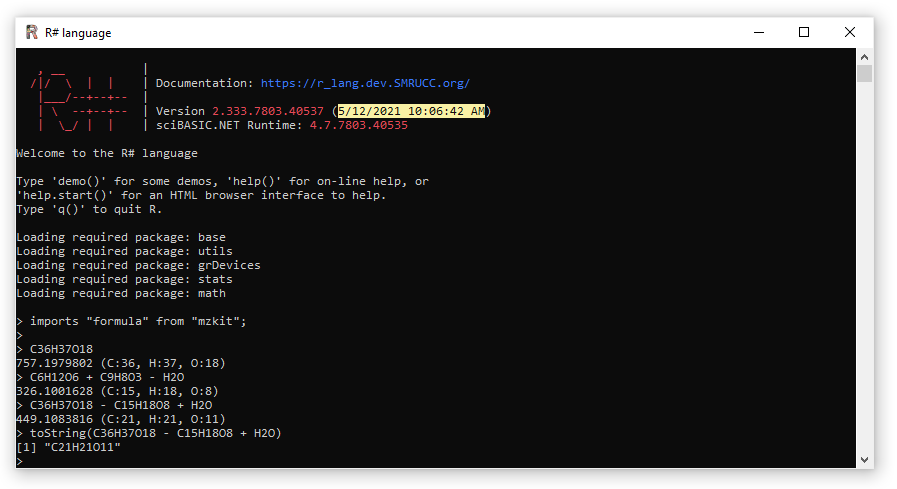
估计阅读时长: 4 分钟根据工作的需要,我为R#脚本解释器添加了一个符号计算的功能,这个符号语言特性在进行一些化学信息学分析的时候会非常有用。例如,我们在分析一些天然产物的质谱数据的时候,会需要通过母离子减掉一些糖来进行中性丢失的计算,基于中性丢失计算来进行一些解谱分析操作。在这个过程之中,化学式符号计算就可以派上很大用场了。假设我们有一个天然产物Cyanidin 3-glucoside-5-(6-p-coumaroylglucoside),从名称我们就可以看出这个天然产物是由一个Cyanidin母核,加上两个葡萄糖以及一个coumaroyl基团构成。这个天然产物的分子化学式为C36H37O18,那现在我们将这个化学式输入到R#解释器之中,按下回车就可以很清楚的了解到这个化学式的元素构成 > C36H37O18 757.1979802 (C:36, H:37, O:18) 如果想要使用这个特性,需要在R#终端上导入mzkit程序包模块:imports "formula" from "mzkit"; Order by Date Name […]
Posts in chemical
© 2025 この中二病に爆焔を!. Created for free using WordPress and
Colibri


Hello There. I found your website through the usage of msn. This is a very smartly written article. I will…
[…] […]
[…] […]
[…] 《为大语言模型运行添加工具调用》 […]
[…] 《从头创建一个DeepSeek客户端》 […]