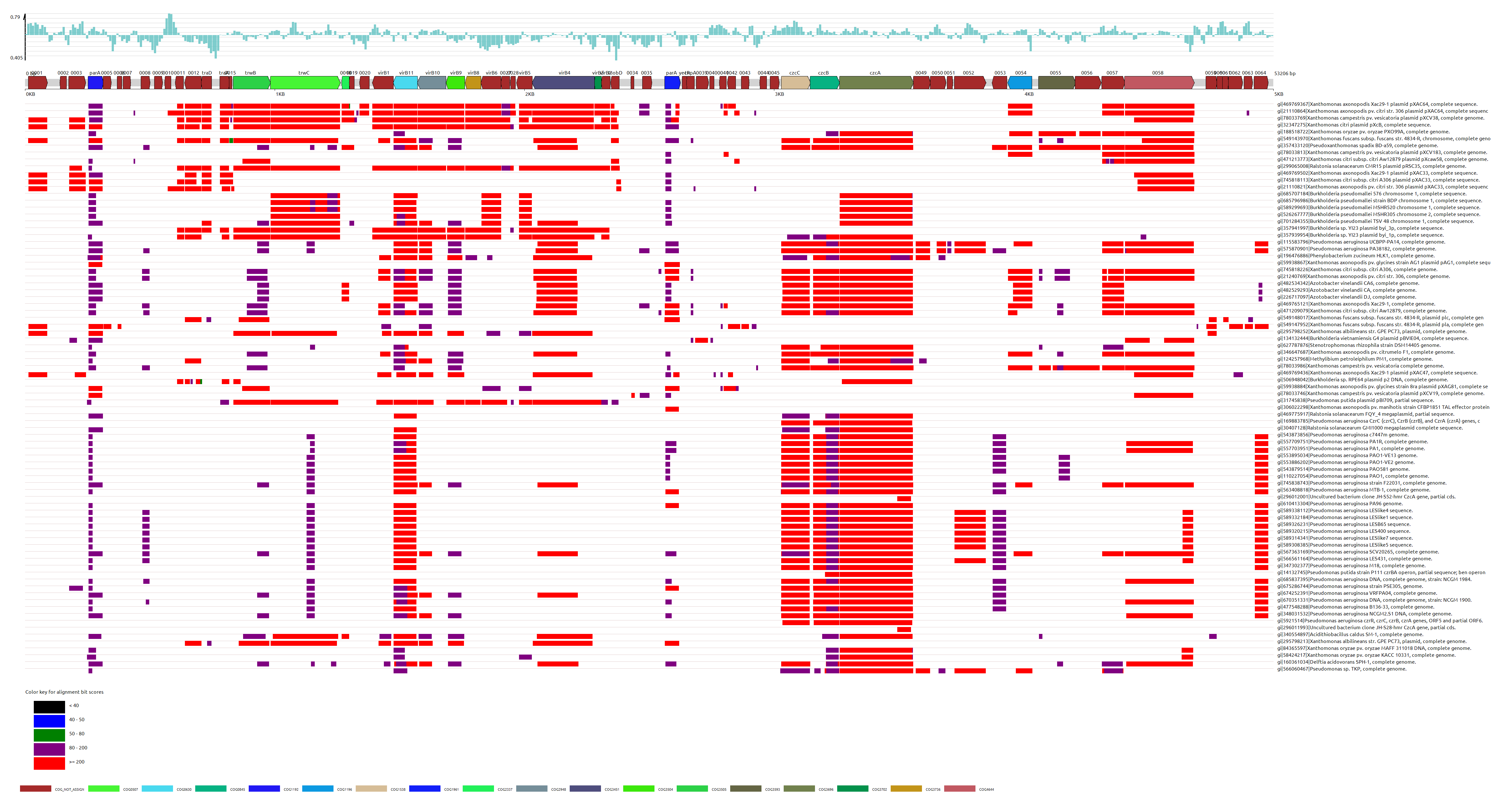
估计阅读时长: 16 分钟KEGG 里面目前并没有“现成的每个 KO 一条代表性序列 FASTA”这种官方序列数据库,假若我们需要基于KEGG数据库中的KO信息的注释,那我们一般会需要自己从 KEGG GENES 里面把每个 KO 对应的基因/蛋白序列抓出来,再按 KO 编号组织成 fasta 集合构建出对应的数据库。基于所建立好的KEGG基因序列数据库,我们就可以实现下面的一些基因注释工作: 在全基因组规模代谢网络重建工作中,进行我们的目标基因组中的代谢网络中的酶节点的直系同源推断,从而将我们的目标基因组中的基因映射到具体的KEGG代谢网络上的节点位置,从而重建出代谢网络模型(使用带有KO编号的蛋白序列做比对注释) 假若我们在进行宏基因组的基因丰度的计算,则可以基于所建立的KEGG基因序列数据库作为参考库,进行宏基因组测序数据中的KO基因丰度的计算(使用带有KO编号的基因序列做比对注释) […]
估计阅读时长: 34 分钟在前面写了一篇文章来介绍我们可以如何通过KEGG的BHR评分来注释直系同源。在KEGG数据库的同源注释算法中,BHR的核心思想是“双向最佳命中”。它比简单的单向BLAST搜索(例如,只看你的基因A在数据库里的最佳匹配是基因B)更为严格和可靠。在基因注释中,这种方法可以有效减少因基因家族扩张、结构域保守等原因导致的假阳性注释,从而更准确地识别直系同源基因,而直系同源基因通常具有相同的功能。在今天重新翻看了下KAAS的帮助文档之后,发现KAAS系统中更新了下面的Assignment score计算公式: We define a score for each ortholog group in order to assign the best […]
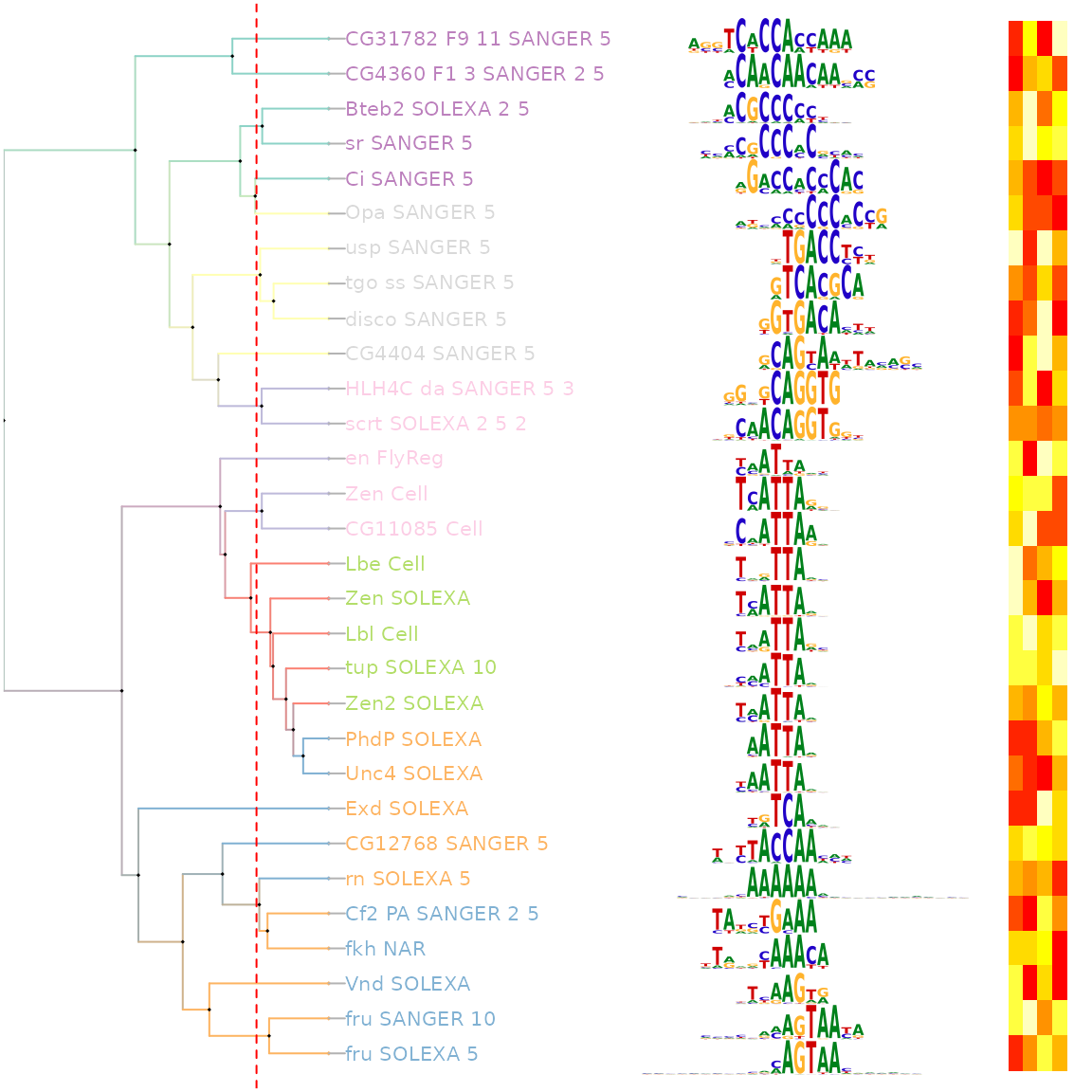
估计阅读时长: 7 分钟一般而言,进行全基因组的转录表达调控网络的建立,我们需要基于两个数据结果来完成: 目标基因的转录调控位点信息(Motif搜索结果,构成网络之中的节点) 转录调控位点相应的转录调控因子(Motif位点相关的转录调控因子,构成网络之中的边连接) Order by Date Name Attachments Xor • 271 kB • 539 click 2022年6月11日An […]
估计阅读时长: 12 分钟Motif是一段典型的序列或者一个结构。一般情况下是指构成任何一种特征序列的基本结构。通俗来讲,即是有特征的短序列,一般认为它是拥有生物学功能的保守序列,可能包含特异性的结合位点,或者是涉及某一个特定生物学过程的有共性的序列区段。比如蛋白质的序列特异性结合位点,如核酸酶和转录因子。 Order by Date Name Attachments Smith-Waterman-Algorithm-Example-Step3 • 8 kB • 531 click 2022年6月7日motifPilesHeatmap-1 • 227 […]
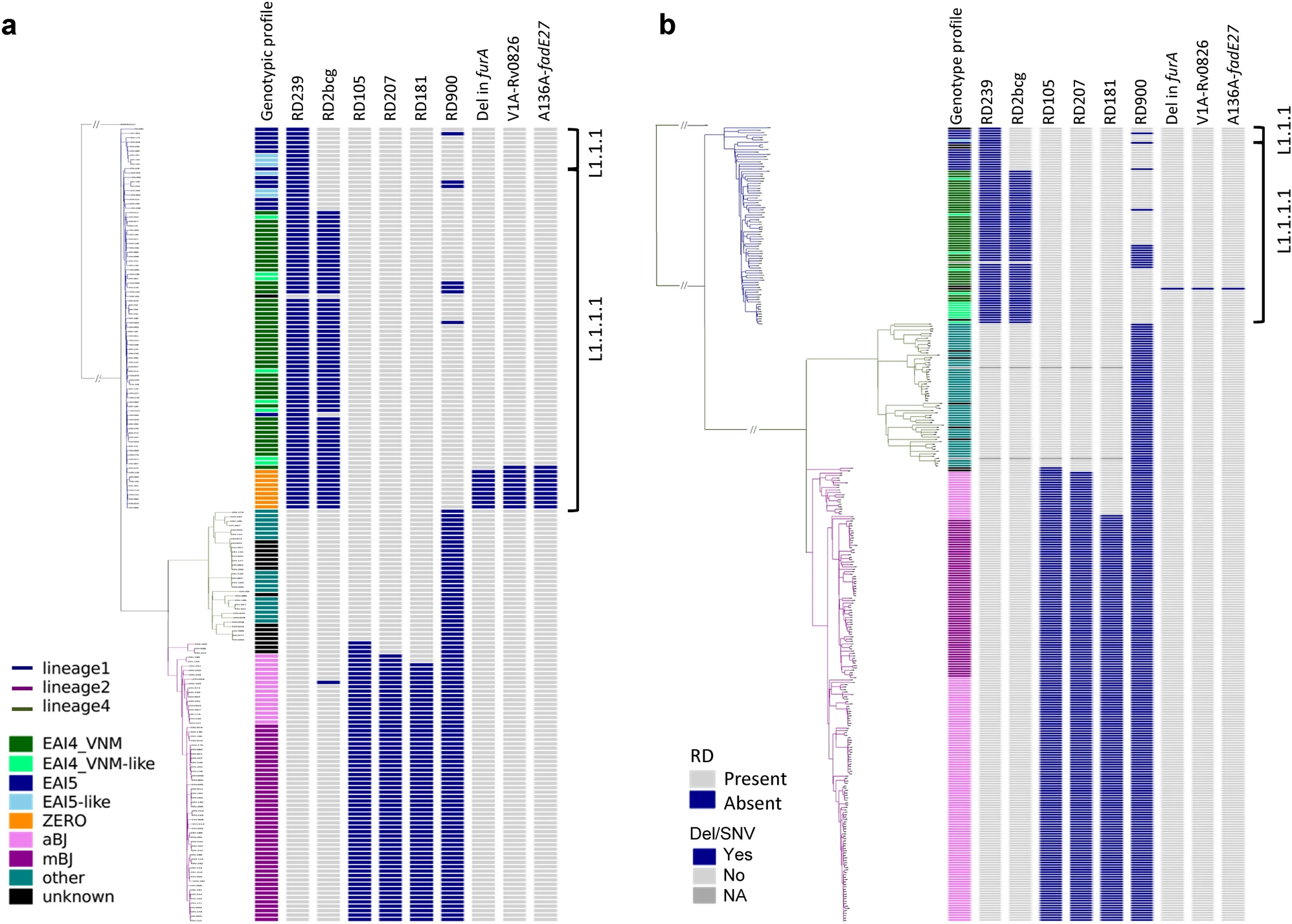
估计阅读时长: 7 分钟F统计量是群体遗传学中由Sewall Wright提出的重要统计量,用于衡量遗传变异在群体中的分布情况。它提供了对群体遗传结构和遗传分化的定量描述。F统计量主要有三种类型:Fis、Fit和Fst,分别反映个体内的、总体的和群体间的遗传分化。F统计量在群体遗传学中通常指的是Fst(Fixation Index,固定指数),它是一个衡量群体间遗传差异的指标。Fst的值范围从0到1,其中0表示群体间没有差异,1表示群体间完全分离。在群体遗传学研究中,Fst常用于评估群体的遗传多样性、群体间的迁移率以及自然选择的压力等。 Order by Date Name Attachments 41598_2021_92984_Fig1_HTML • 2 MB • 581 click 2022年5月28日p1 […]
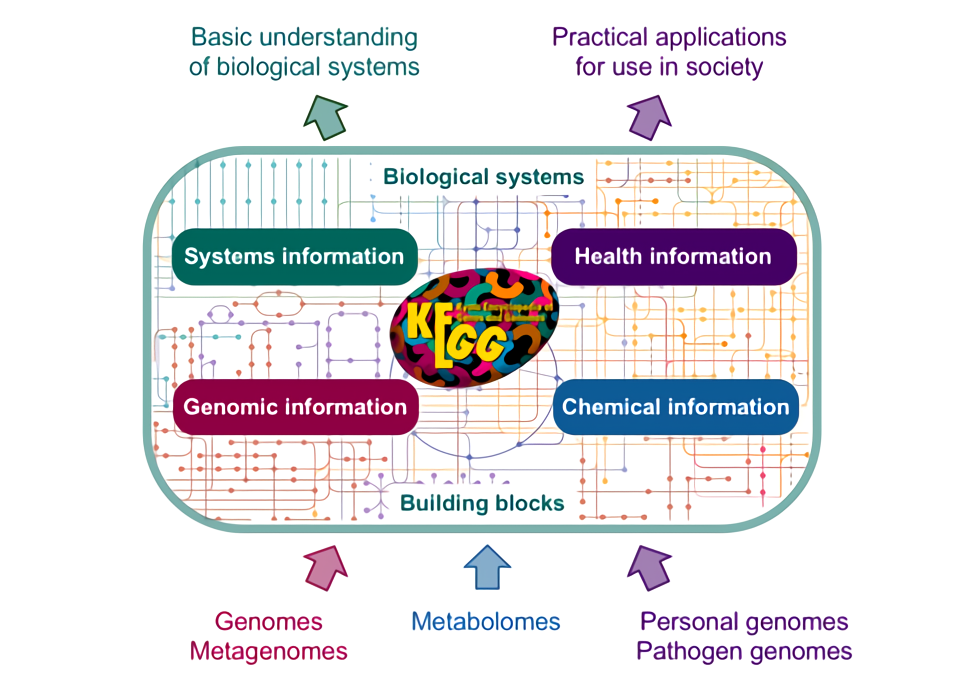
估计阅读时长: 14 分钟在基因组学研究中,将新测序的基因或者针对目标基因组进行基于KEGG代谢通路体系的虚拟细胞建模,都会需要将目标基因组与已知功能基因进行比对注释。KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库通过其KEGG Orthology (KO)系统,为基因功能注释提供了一个强大的平台。KO系统将功能上保守的直系同源基因归为一类,每个KO条目(K编号)代表一个直系同源基因群,这些基因在不同物种中通常执行相似的生物学功能。因此,将新基因的序列与KEGG数据库中的已知基因进行比对,可以推断其可能的KO编号,从而将其功能映射到KEGG通路图或功能层级中。 Order by Date Name Attachments kegg_overview • 313 […]



Hi, appreciate the effort put into this. It's always good to see quality content. 🥳
WOOOOOW This was incredibly helpful and easy to understand. I've learned a lot. many thanks to your idea sharing.
[…] 在前面写了一篇文章来介绍我们可以如何通过KEGG的BHR评分来注释直系同源。在KEGG数据库的同源注释算法中,BHR的核心思想是“双向最佳命中”。它比简单的单向BLAST搜索(例如,只看你的基因A在数据库里的最佳匹配是基因B)更为严格和可靠。在基因注释中,这种方法可以有效减少因基因家族扩张、结构域保守等原因导致的假阳性注释,从而更准确地识别直系同源基因,而直系同源基因通常具有相同的功能。在今天重新翻看了下KAAS的帮助文档之后,发现KAAS系统中更新了下面的Assignment score计算公式: […]
不常看到, 没有多余矫饰的表达。敬意。
[…] 在前面写了一篇文章来介绍我们可以如何通过KEGG的BHR评分来注释直系同源。在今天重新翻看了下KAAS的帮助文档之后,发现KAAS系统中更新了下面的Assignment score计算公式: […]