文章阅读目录大纲
Connected Component Labeling(连通组件标记算法)主要用于识别并标记二值图像中相互连接的像素区域(即连通区域)。
imports "geometry2D" from "graphics";
imports "machineVision" from "signalKit";
let raw = readImage("—Pngtree—five chickens in different colors_3632916.jpg");
let bin = machineVision::ostu(raw, factor = 0.8);
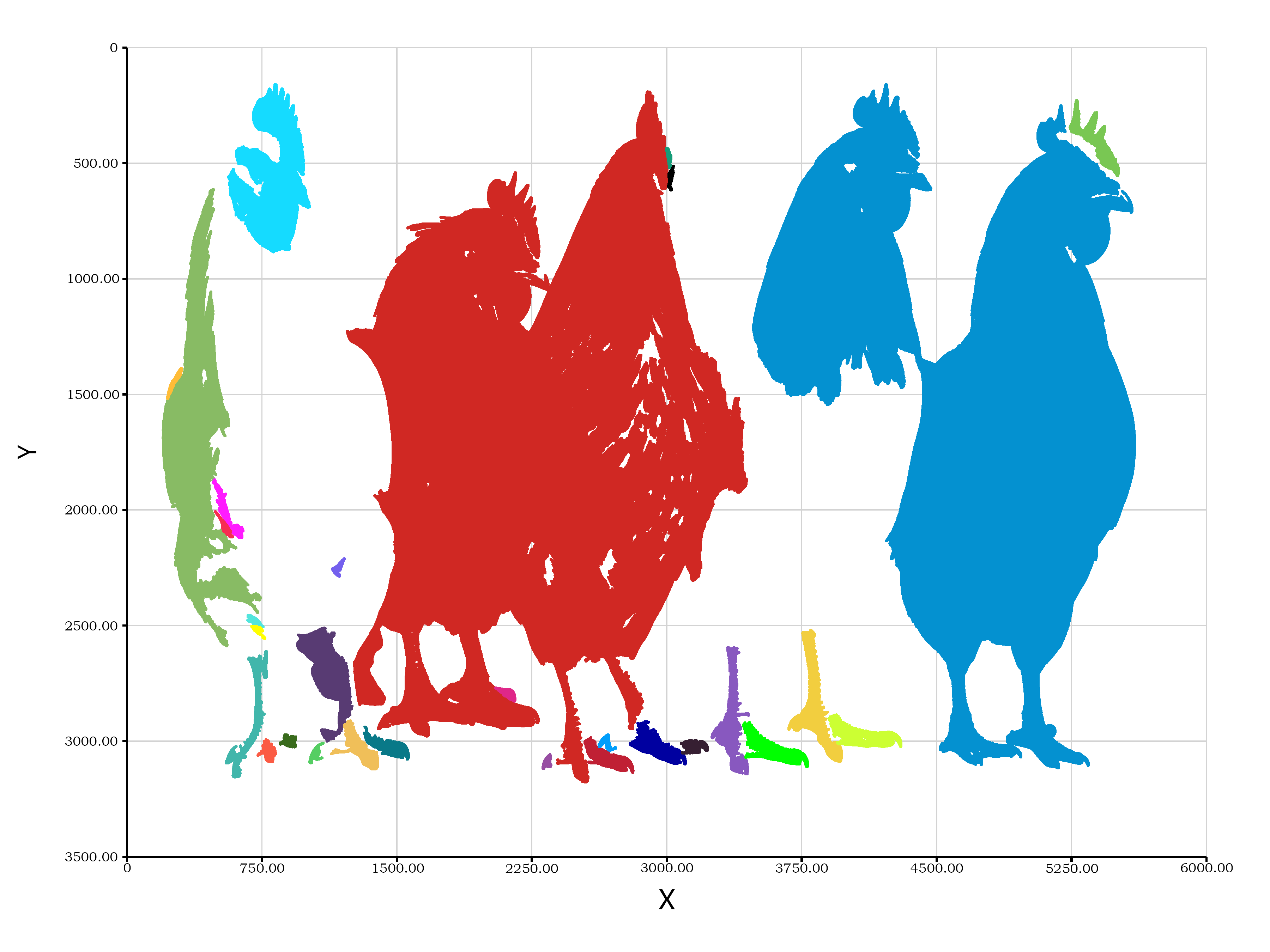
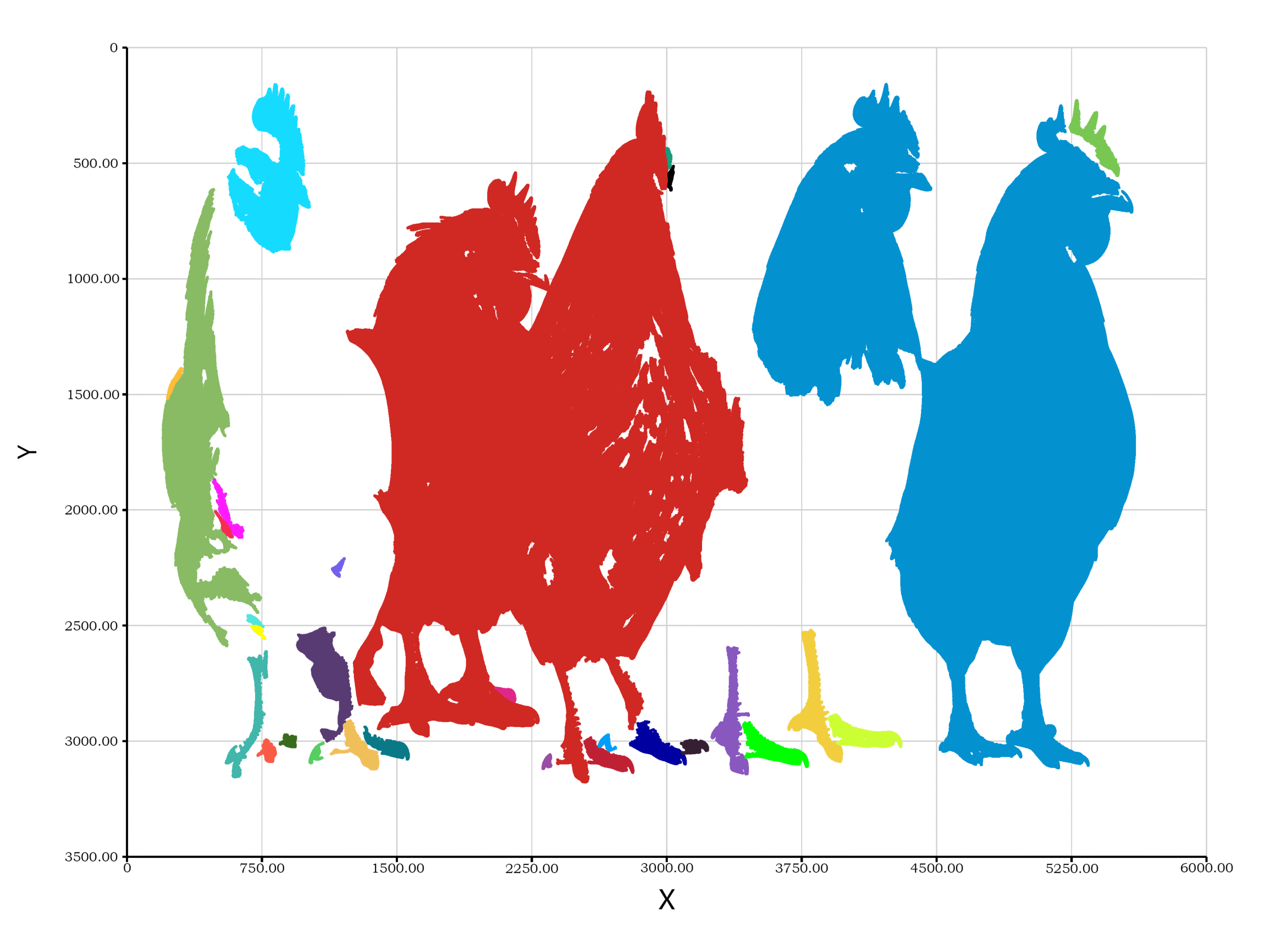
let shapes = machineVision::ccl(bin);
print(`find ${length(shapes)} shapes.`);
bitmap(bin, file = "ostu_bin.bmp");
bitmap(file = "shapes.png", size = [3600, 2700]) {
plot(shapes, scatter = TRUE, padding = "padding: 5% 5% 10% 10%;");
}

Latest posts by 谢桂纲 (see all)
- 酶EC编号结构解析 - 2026年2月17日
- 二叉树聚类可视化微生物群落代谢差异 - 2026年2月15日
- 通过diamond软件进行blastp搜索 - 2026年2月15日


6 Responses
Je pense que cet algorithme présente encore des limitations importantes. Par exemple, sur plusieurs poules présentes sur l’image originale, l’une d’elles, parce qu’elle a des plumes blanches, voit ces dernières devenir indiscernables du fond blanc après que l’image a été traitée par binarisation de l’algorithme. Cela signifie que les informations caractéristiques de ces plumes ont directement disparu de l’image. C’est la raison pour laquelle, après identification, le corps de la première et de la troisième poule sur l’image apparaît dans un état fragmenté. Si nous pouvions modifier cet algorithme pour qu’il effectue l’analyse et l’identification via une image en niveaux de gris plutôt qu’une image binaire noire et blanche, je pense que les résultats seraient bien meilleurs.
I would like to gain a more detailed understanding of the computational principles behind this algorithm. Could you please provide a more in-depth explanation specifically on this topic?
有两只挨着的母鸡在二值化之后就分不开了
CCL算法没法处理这种连接的对象有这个缺点
This algorithm cannot help me count how many hens there are in the picture.
The algorithm failed to achieve the expected results, primarily because the color details of some hens were not preserved in the binarized image, lacking sufficient information to distinguish them.