估计阅读时长: 5 分钟基因组 为了能够实现上面所描述的这种基于EC Number的不同层级的加权嵌入,我们在原来的基因组嵌入工具上添加了一个hierarchical选项,用于支持切换为层级嵌入的操作: Imports Microsoft.VisualBasic.Data.Framework Imports Microsoft.VisualBasic.Data.NLP Imports SMRUCC.genomics.Interops.NCBI.Extensions.Pipeline Public Class GenomeMetabolicEmbedding ReadOnly vec As New […]
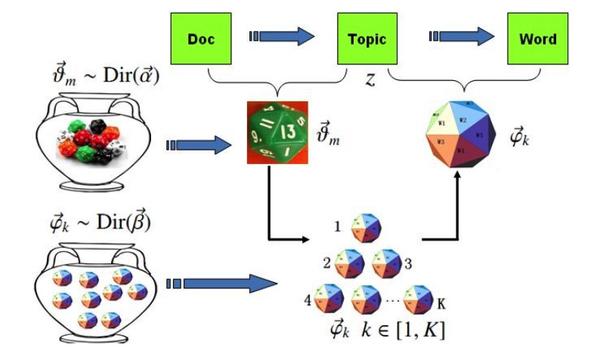
估计阅读时长: 20 分钟LDA(Latent Dirichlet Allocation,潜在狄利克雷分配)是一种用于发现文档集合中潜在主题的生成式概率模型。它假设文档是由多个主题混合而成的,而每个主题又是通过一定的概率分布选择词语生成的。LDA模型包含词、主题和文档三层结构,通过概率生成过程模拟文档的形成。Gibbs LDA 的核心在于使用吉布斯采样方法来推断这些隐藏的主题分布。 Attachments v2-883ac9db7f1cbd7325b2450cd225a897_b • 29 kB • 100 click 2026年2月23日
估计阅读时长: 17 分钟EC Number是国际酶学委员会(IUBMB)制定的一套酶分类编号体系,EC Number采用层级分类法,由4个数字组成,分别代表酶的大类、亚类、亚亚类和序号。例如,“EC 1.1.1.37”中,第一个“1”表示氧化还原酶大类;第二个“1”表示作用于CH-OH基团;第三个“1”表示以NAD+或NADP+为受体的酶;第四个“37”表示特定酶苹果酸脱氢酶。这种层次结构意味着EC编号蕴含了丰富的功能信息,包括酶催化的反应类型和底物/机制。将EC Number嵌入为向量,有助于我们利用机器学习模型进行功能预测、相似性分析等。 Order by Date Name Attachments Capture • 14 kB • 154 […]
估计阅读时长: 11 分钟在将生物序列(如基因组或蛋白质序列)或文本数据转换为数值向量形式时,TF-IDF(Term Frequency-Inverse Document Frequency)和N-gram One-hot(又称Bag-of-n-grams)是两种经典且基础的文档嵌入算法。它们各自侧重于不同的特征提取方式,常被用于自然语言处理和生物信息学领域。 Attachments scatter_plot • 433 kB • 153 click 2026年2月10日
估计阅读时长: 24 分钟假若现在有两条Fasta序列放在你面前,现在需要你进行这两条Fasta序列的相似度计算分析。如果对于我而言,大学刚毕业刚入门生物信息学的时候,可能只能够想到通过blast比对的方式进行序列相似性计算分析。基于blast比对方式可以找到生物学意义上的序列相似性结果,但是计算的效率会比较低。假设现在让你使用这些序列进行机器学习建模分析,或者基于传统数学意义上的基于相似度的无监督聚类分析的时候,面对这些长度上长短不一的生物序列数据,可能会比较蒙圈,因为传统的数学分析方法都要求我们分析的目标至少应该是等长的向量数据。 Order by Date Name Attachments Fasta-A • 544 kB • 630 click 2023年6月29日visualize • 45 […]
Recent Posts
Archives
- February 2026 (9)
- January 2026 (2)
- December 2025 (10)
- November 2025 (2)
- October 2025 (1)
- August 2025 (3)
- July 2025 (2)
- June 2025 (6)
- May 2025 (3)
- November 2023 (1)
- June 2023 (2)
- May 2023 (2)
- April 2023 (2)
- March 2023 (2)
- February 2023 (1)
- August 2022 (2)
- July 2022 (2)
- June 2022 (5)
- May 2022 (5)
- April 2022 (4)
- March 2022 (3)
- January 2022 (2)
- December 2021 (2)
- November 2021 (2)
- October 2021 (6)
- September 2021 (8)
- August 2021 (8)
- July 2021 (6)
- June 2021 (20)
- May 2021 (10)
博客文章
| S | M | T | W | T | F | S |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 |
| 8 | 9 | 10 | 11 | 12 | 13 | 14 |
| 15 | 16 | 17 | 18 | 19 | 20 | 21 |
| 22 | 23 | 24 | 25 | 26 | 27 | 28 |
| 29 | 30 | 31 | ||||
Tags
algorithm (33)
bilibili (3)
binary tree (4)
clustering (21)
Darwinism (4)
data visualization (23)
dotnet-core (25)
ec number (4)
embedding (5)
GCModeller (20)
gdi+ (23)
gem (7)
ggplot (14)
graph (14)
heatmap (5)
http (4)
image processing (7)
kegg (8)
kmeans (3)
language (7)
linq (3)
linux (8)
machine learning (4)
mass spectrometry (12)
math (19)
metagenomics (6)
motif (4)
MSI (4)
mzkit (19)
network (8)
pathway (4)
pipeline (4)
query (5)
R# (44)
rsharp (23)
scripting (14)
single-cell (6)
sql (3)
symbolic computation (3)
text processing (4)
typescript (3)
ubuntu (4)
uniprot (3)
vb (19)
VisualBasic (50)

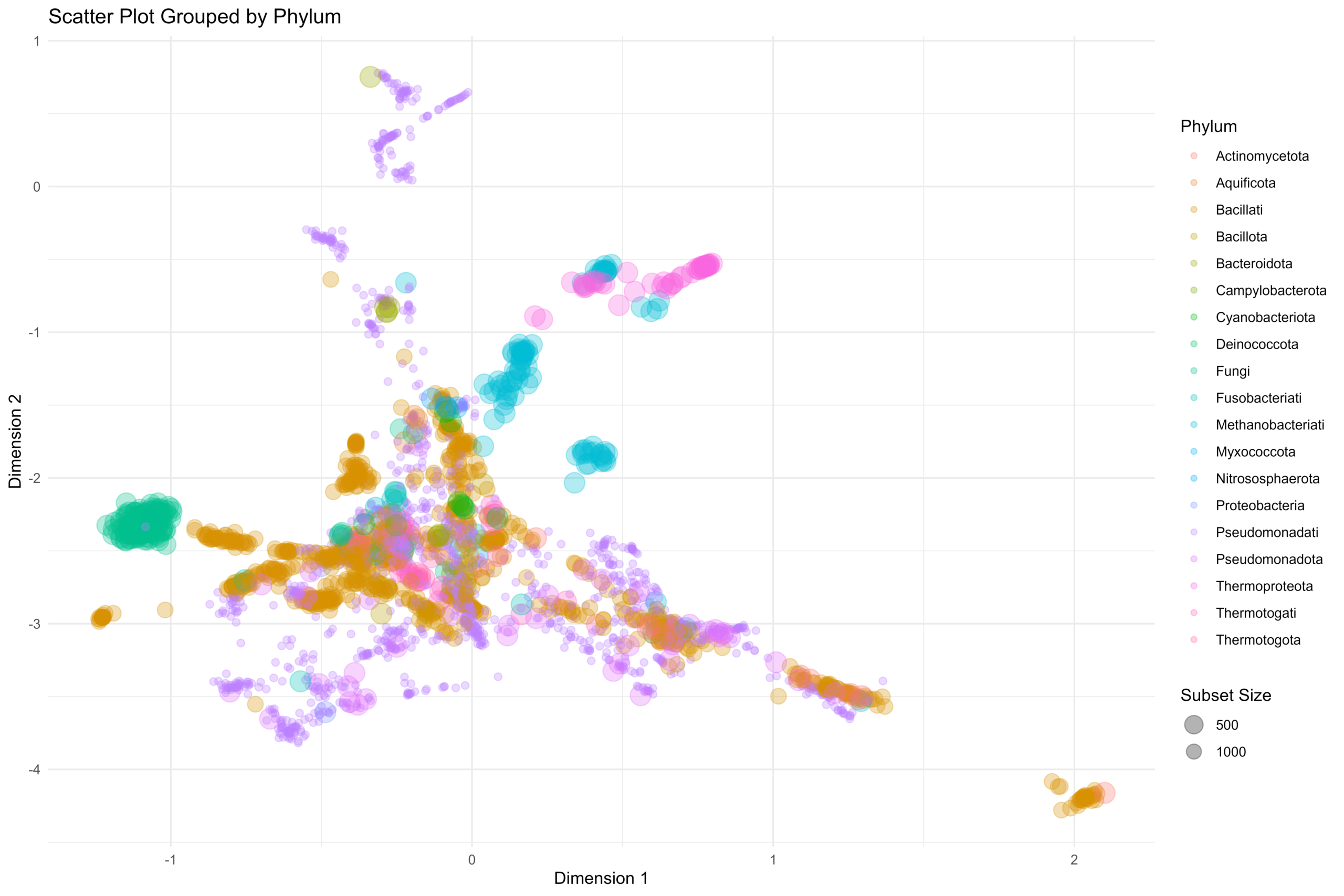
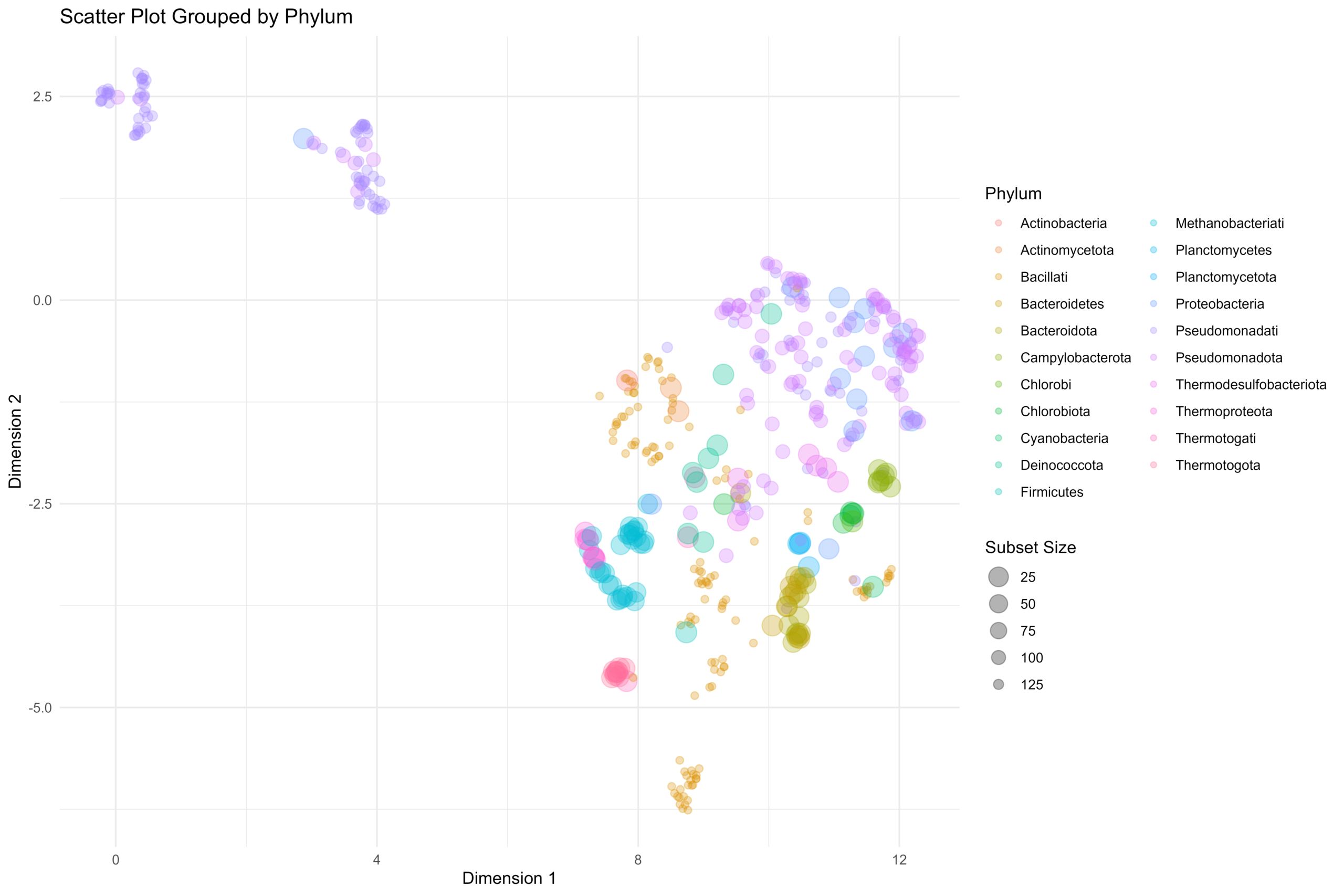


[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 对于基于ec number来生成层级数据,我们直接使用《酶EC编号结构解析》文章末尾所展示的层级数据生成函数来实现。 […]
[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?