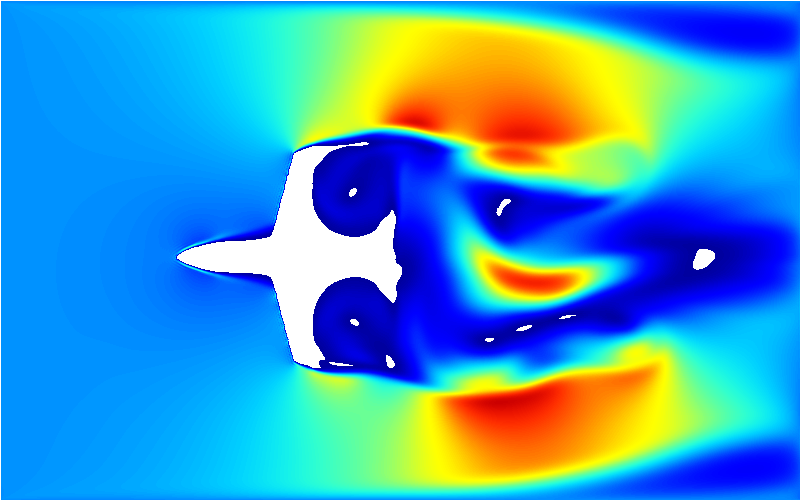
估计阅读时长: 30 分钟https://github.com/xieguigang/Moira LBM(格子玻尔兹曼方法)凭借其介观模型特性,在流体模拟领域展现出显著技术优势:其碰撞与迁移过程仅依赖局部数据,天然适配GPU并行计算,CUDA实现可达成10–100倍加速比;处理复杂几何边界时无需生成体网格,通过格点标记固体并配合反弹边界即可高效实现,尤其适用于多孔介质等场景;同时,通过扩展分布函数可灵活耦合多物理场,例如引入温度分布函数模拟传热,或采用伪势模型捕获多相流中的相分离现象。尽管在高速或高粘度流动中存在局限,但通过MRT算法优化及GPU硬件加速,LBM已成为微流动、多孔介质、多相流等复杂流体模拟的理想工具,在航空工程等领域已有成功应用案例,其应用前景持续拓展。 Order by Date Name Attachments frame-00093 • 2 MB • 530 click 2025年8月9日ffmpeg • […]
Posts in 仿真模拟
© 2026 この中二病に爆焔を!. Created for free using WordPress and
Colibri


[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 对于基于ec number来生成层级数据,我们直接使用《酶EC编号结构解析》文章末尾所展示的层级数据生成函数来实现。 […]
[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?