估计阅读时长: 9 分钟https://github.com/rsharp-lang/Rnb 之前使用Python脚本进行编写代码的时候,十分的羡慕Python脚本可以基于ipynb记事本进行文档化的编码。在之前R#脚本是缺少相关的代码库模块将可执行的R#脚本渲染成可视化文档。但是经过几天的开发工作时候,现在R#脚本编程已经具备有了文档化编程的基本框架了。 Order by Date Name Attachments 01510007-school-notebook • 32 kB • 669 click 2021年10月30日renderHtml_cli • […]
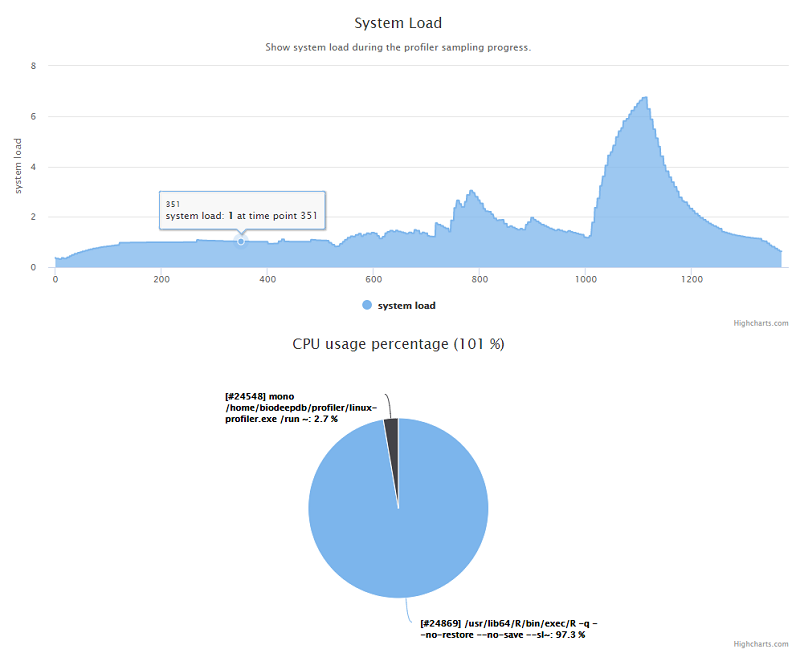
估计阅读时长: 6 分钟https://github.com/xieguigang/linux-profiler 废话不多说,首先给出一个 demo报告链接 给大家看看这个小工具的成品输出。 在去年的工作中,因为公司需要购买新的服务器做集群计算,需要一个工具来记录之前的服务器在数据分析上的性能瓶颈。于是花了两天的时间赶出来了这个专门应用于Linux系统的性能记录工具。这个小工具是一个开源项目,大家可以在Github上阅读这个开源项目(linux-profiler)的源代码。 Order by Date Name Attachments systemLoad • 53 kB • 754 […]
估计阅读时长: < 1 分钟Welcome to WordPress. This is your first post. Edit or delete it, then start writing!



[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 对于基于ec number来生成层级数据,我们直接使用《酶EC编号结构解析》文章末尾所展示的层级数据生成函数来实现。 […]
[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?