估计阅读时长: 7 分钟https://github.com/xieguigang/mzkit SMILES字符串是一种在计算化学领域内使用线性ASCII字符串描述一个具有空间立体结构的分子结构所使用的一种语言规范。因为在工作中会需要使用到SMILES字符串做一些分子结构相关的数据建模分析,所以编写了一个很方便的用于SMILES字符串解析操作的模块,在这篇文章中为大家讲解具体的工作原理。 Order by Date Name Attachments science-connection-structure-with-molecules-simple-modern-white-background-illustration_46577-719 • 36 kB • 760 click 2021年6月9日abstract-molecules-structure-with-connect-spherical-particles_46577-689 • […]
估计阅读时长: 2 分钟https://github.com/xieguigang/mzkit 在BILIBILI上观看视频:《【BioNovoGene Mzkit教程】代谢组学原始数据处理基础》 Order by Date Name Attachments profile_videocard • 211 kB • 766 click 2021年5月29日metabolims […]
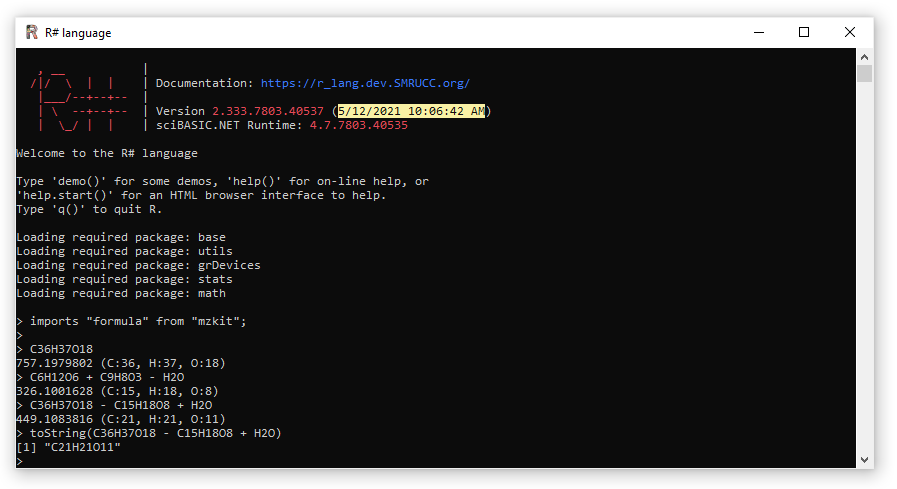
估计阅读时长: 4 分钟根据工作的需要,我为R#脚本解释器添加了一个符号计算的功能,这个符号语言特性在进行一些化学信息学分析的时候会非常有用。例如,我们在分析一些天然产物的质谱数据的时候,会需要通过母离子减掉一些糖来进行中性丢失的计算,基于中性丢失计算来进行一些解谱分析操作。在这个过程之中,化学式符号计算就可以派上很大用场了。假设我们有一个天然产物Cyanidin 3-glucoside-5-(6-p-coumaroylglucoside),从名称我们就可以看出这个天然产物是由一个Cyanidin母核,加上两个葡萄糖以及一个coumaroyl基团构成。这个天然产物的分子化学式为C36H37O18,那现在我们将这个化学式输入到R#解释器之中,按下回车就可以很清楚的了解到这个化学式的元素构成 > C36H37O18 757.1979802 (C:36, H:37, O:18) 如果想要使用这个特性,需要在R#终端上导入mzkit程序包模块:imports "formula" from "mzkit"; Order by Date Name […]




[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 对于基于ec number来生成层级数据,我们直接使用《酶EC编号结构解析》文章末尾所展示的层级数据生成函数来实现。 […]
[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?