文章阅读目录大纲
在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。除了针对降维后的数据进行散点图可视化,我们还可以直接针对向量化嵌入后的原始嵌入矩阵进行聚类,完成聚类结果的可视化。在这里我们主要是基于嵌入的原始结果进行二叉树聚类可视化。
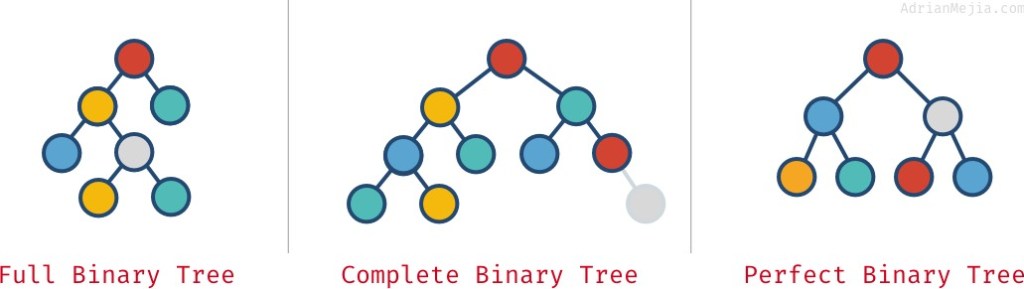
二叉树聚类原理讲解
所谓的二叉树聚类,指的就是我们基于嵌入的结果向量进行基于相似度的二叉树构建的过程。假设我们将构建出来的基因组代谢酶注释模型看作为二叉树上的某一个节点,在构建出一颗二叉树的时候会需要计算新插入的基因组数据和二叉树上已经存在的节点中的基因组数据之间的相似度,在完成了相似度的计算操作之后,我们会按照相似度阈值情况进行下面的操作:
- 假设相似度小于某一个阈值,例如小于0.8,则认为当前需要插入到树上的基因组信息和当前所比较的二叉树节点完全不一样,这个时候会将这个基因组数据往节点的左边插入,然后递归的往后计算
- 假设相似度大于某一个阈值,例如大于等于0.8,则认为当前需要插入到树上的基因组信息和当前所比较的二叉树节点有很高的相似度,这个时候会将这个基因组数据往节点的右边插入,然后递归的往后计算
- 更进一步的,假设相似度大于某一个特定的阈值,例如大于等于0.95,则认为当前需要插入到数据的基因组信息和当前所比较的二叉树节点上的基因组信息完全一致,则我们直接把当前的基因组信息追加到当前所比较的二叉树节点上。
这样子,基于第三个步骤的比较,假若有5个基因组的注释结果信息和当前的二叉树的基因组注释结果信息相比较,都是大于所设定的最高的特定阈值的,则当前的二叉树节点上将会匹配有6个基因组的注释结果信息。按照这样子的原理,就可以实现基于二叉树的聚类方法了。
二叉树聚类与进化树的差异
对二叉树聚类的结果我们可以很容易的想象得出,我们基于基因组的嵌入结果向量之间的相似度,建立起了一棵树。而基于这样子的矩阵信息所构建出一棵树的方法还有层次聚类方法。假若我们使用平均距离加权的方法构建出一颗层次聚类树,那么实际上我们就是基于UPGMA算法构建了一颗进化树了。在下面的R#示例脚本代码中,基于前面我们所建立的基因组代谢酶注释向量化嵌入的结果,实现了UPGMA算法的层次聚类树的构建,并进行了相应的进化树的绘制:
require(GCModeller);
require(umap);
imports "clustering" from "MLkit";
imports "taxonomy_kit" from "metagenomics_kit";
# read metabolic embedding data
let metabolic_vec = read.csv("metabolic_embedding.csv", row.names = 1, check.names = FALSE);
# parse the taxonomy class label in phylum rank level
let taxon = biom_string.parse(metabolic_vec$taxonomy) |> taxonomy_name(rank = "phylum");
metabolic_vec[,"taxonomy"] = NULL;
rownames(metabolic_vec) = unique.names(taxon);
# create color mapping
let colors = as.list(palette(taxon, "paper"), names = rownames(metabolic_vec));
let latent = umap(metabolic_vec, dimension = 9, numberOfNeighbors =128, localConnectivity = 2, method="Cosine");
# build UPGMA tree
let dtree = as.data.frame(latent$umap, labels = latent$labels)
|> dist()
|> hclust(method = "average")
;
# make plot visual of the cluster tree
bitmap(file = "./hclust_samples.png", size = [3600, 5000]) {
plot(dtree,
class = colors,
padding = "padding: 150px 100px 100px 100px;",
axis.format = "G3",
links = "stroke: gray; stroke-width: 2px; stroke-dash: dash;",
pt.color = "lightgray",
label = "font-style: normal; font-size: 8; font-family: Microsoft YaHei;",
ticks = "font-style: normal; font-size: 20; font-family: Microsoft YaHei;",
pt.size = 2
);
}与二叉树聚类方法所不同的是,我们在上面进行UPGMA层次聚类树的构建的时候,使用的是欧几里得距离进行基因组之间的距离的计算,得到的距离结果值越大,说明所计算的两个基因组之间的差异越大。而在二叉树聚类中,我们则是通过cosine相似度进行二叉树的构建,得到的相似度结果值越大,说明所计算的两个基因组之间的差异越小,相似度越高。在比较两个基因组之间的差异这个metric上,UPGMA算法和这里讨论的二叉树聚类方法就很明显不一样。
与UPGMA算法的另一个不同点就是,二叉树聚类是没有根的,或者说最终得到的二叉树的形状是由根节点所决定的。由于我们在进行二叉树聚类的时候,所输入的基因组数据之间的顺序可能是随机的,所以不同顺序的基因组数据的输入,得到的二叉树的形状也就不大相同。对于UPGMA算法层次聚类树而言,则不会受到这个问题的限制,其总是会得到一个形状固定的有根的树。
运行上面的脚本代码,我们得到了下面的UPGMA层次聚类树:

二叉聚类树实现代码
下面,我们就来了解我们是如何实现出计算基因组代谢注释嵌入结果向量为二叉树结果的过程。
基因组节点相似度比较的实现
在基础的算法模块之中,有下面的一个抽象对象用于定义两个对象的相似度比较的计算过程。从下面的代码中可以看得到,程序模块通过GetSimilarity函数,从两个对象之间计算出一个double类型的相似度结果值,然后Compares函数会调用我们所定义GetSimilarity函数,基于这个函数所计算出来的相似度结果值,按照我们所定义的阈值返回两个对象的比较结果。
Imports System.Runtime.CompilerServices
Public MustInherit Class ComparisonProvider
Protected ReadOnly equalsDbl As Double
Protected ReadOnly gt As Double
''' <summary>
''' create a new score generator
''' </summary>
''' <param name="equals">score level for construct a binary tree cluster</param>
''' <param name="gt">score level for create a binary tree branch</param>
Sub New(equals#, gt#)
Me.equalsDbl = equals
Me.gt = gt
End Sub
Public MustOverride Function GetSimilarity(x As String, y As String) As Double
Public MustOverride Function GetObject(id As String) As Object
''' <summary>
''' binary tree generator
''' </summary>
''' <param name="x"></param>
''' <param name="y"></param>
''' <returns></returns>
Public Function Compares(x As String, y As String) As Integer
Dim similarity As Double = GetSimilarity(x, y)
If similarity >= equalsDbl Then
Return 0
ElseIf similarity >= gt Then
Return 1
Else
Return -1
End If
End Function
<MethodImpl(MethodImplOptions.AggressiveInlining)>
Public Function GetComparer() As Comparison(Of String)
Return Me
End Function
<MethodImpl(MethodImplOptions.AggressiveInlining)>
Public Shared Narrowing Operator CType(c As ComparisonProvider) As Comparison(Of String)
Return New Comparison(Of String)(AddressOf c.Compares)
End Operator
End Class
' module for compares the similarity between two genome metabolic model
Public Class OTUCosineComparer : Inherits ComparisonProvider
ReadOnly OTUs As New Dictionary(Of String, OTUTable)
ReadOnly sampleids As String()
Public ReadOnly Property OTU_ids As IEnumerable(Of String)
Get
Return OTUs.Keys
End Get
End Property
Public Sub New(OTUs As IEnumerable(Of OTUTable), equals As Double, gt As Double)
MyBase.New(equals, gt)
For Each otu As OTUTable In OTUs.SafeQuery
Call Me.OTUs.Add(otu.ID, otu)
Next
sampleids = Me.OTUs.Values.PropertyNames
End Sub
Public Overrides Function GetSimilarity(x As String, y As String) As Double
Dim otu1 As OTUTable = OTUs(x)
Dim otu2 As OTUTable = OTUs(y)
Dim P As Double() = otu1(sampleids)
Dim Q As Double() = otu2(sampleids)
Return Cosine.SSM_SIMD(P, Q)
End Function
Public Overrides Function GetObject(id As String) As Object
Return OTUs(id)
End Function
End Class在上面的比较代码中,similarity >= equalsDbl所实现的就是我们的最高一级的比较,超过equalsDbl这个阈值就意味着两个比较的对象是一致的,则这个时候函数返回零,表示将当前的基因组信息插入到当前的二叉树节点上;当程序比较发现similarity >= gt的时候,就意味着两个基因组的比较结果超过了相似度阈值,但是还没有到完全一致的那种程度,即将当前的基因组信息插入到了当前的二叉树节点的右边。对于其他的没有超过所设定的最低阈值的情况下,函数就返回-1,表示需要将当前的基因组信息插入到当前的二叉树节点的左侧。在所实现的OTUCosineComparer子类中,则是将嵌入向量数据提取出来后,通过计算两个向量之间的cosine相似度来实现GetSimilarity抽象方法。
基于上面的相似度比较器的定义,我们可以完成下面的二叉树的构建代码:
<Extension>
Public Function BTreeCluster(uniqueId As IEnumerable(Of String), alignment As ComparisonProvider) As BTreeCluster
Dim btree As New AVLTree(Of String, String)(alignment.GetComparer, Function(str) str)
For Each id As String In uniqueId
Call btree.Add(id, id, valueReplace:=False)
Next
Return BTreeCluster.GetClusters(btree, alignment)
End Function
Private Function Add(key As K, value As V,
tree As BinaryTree(Of K, V),
callback As DelegateTreeInsertCallback(Of K, V)) As BinaryTree(Of K, V)
If tree Is Nothing Then
' 追加新的叶子节点
tree = New BinaryTree(Of K, V)(key, value, Nothing, views)
stack.Add(tree)
Else
Select Case compares(key, tree.Key)
Case < 0 : Call appendLeft(tree, key, value, callback)
Case > 0 : Call appendRight(tree, key, value, callback)
Case = 0
' 将value追加到附加值中(也可对应重复元素)
Call callback.insertDuplicated(tree, value)
' 2018.3.6
' 如果是需要使用二叉树进行聚类操作,那么等于零的值可能都是同一个簇之中的
' 在这里将这个member添加进来
Call DirectCast(tree!values, List(Of V)).Add(value)
Case Else
Throw New Exception("This will never happend!?")
End Select
End If
Call tree.PutHeight
Return tree
End Function在上面的二叉树构建的代码中片段中,我们是针对基因组的唯一ID进行比较构建。通过向二叉树中插入基因组的唯一ID,然后程序模块通过唯一ID获取得到对应的嵌入后的向量结果,然后基于两个唯一ID得到的两个向量进行cosine相似度的计算,之后就可以根据阈值判断结果为0,1或者-1结果值。这样子最后,在Add函数中就可以按照所计算出来的结果值来将基因组数据选择插入到左边还是右边,或者插入到当前的节点中实现出聚类效果。
在得到了一颗二叉树之后,我们可以按照下面的方法模块,以递归的方式将二叉树提取为网络图对象,以方便进行导出,在cytoscape软件程序中进行可视化。
Imports System.Runtime.CompilerServices
Imports Microsoft.VisualBasic.ComponentModel.Collection
Imports Microsoft.VisualBasic.Data.visualize.Network.FileStream.Generic
Imports Microsoft.VisualBasic.Data.visualize.Network.Graph
Public Module ClusterViz
''' <summary>
'''
''' </summary>
''' <param name="tree"></param>
''' <param name="metadata">
''' there are some special metadata key:
'''
''' 1. text - for node data label
''' 2. value - for node data mass weight
''' </param>
''' <returns></returns>
<Extension>
Public Function MakeTreeGraph(tree As BTreeCluster, Optional metadata As Func(Of String, Dictionary(Of String, String)) = Nothing) As NetworkGraph
Dim g As New NetworkGraph
Call tree.PullTreeGraph(g, If(metadata, EmptyMetadata()))
Return g
End Function
Private Function EmptyMetadata() As Func(Of String, Dictionary(Of String, String))
Return Function(any) New Dictionary(Of String, String)
End Function
<Extension>
Private Sub PullTreeGraph(tree As BTreeCluster, g As NetworkGraph, metadata As Func(Of String, Dictionary(Of String, String)))
Dim root As Node = g.CreateNode(tree.uuid)
root.data.Add(metadata(root.label))
root.data.Add(NamesOf.REFLECTION_ID_MAPPING_NODETYPE, root.label)
root.data.label = root.data.Properties.Popout("text")
root.data.mass = Val(root.data.Properties.Popout("value"))
For Each id As String In tree.members
If id <> root.label Then
Dim v As Node = g.CreateNode(id)
v.data.Add(metadata(id))
v.data.Add(NamesOf.REFLECTION_ID_MAPPING_NODETYPE, root.label)
v.data.label = v.data.Properties.Popout("text")
v.data.mass = Val(v.data.Properties.Popout("value"))
g.CreateEdge(root, v)
End If
Next
If tree.left IsNot Nothing Then
Call tree.left.PullTreeGraph(g, metadata)
Call g.CreateEdge(root, g.GetElementByID(tree.left.uuid))
End If
If tree.right IsNot Nothing Then
Call tree.right.PullTreeGraph(g, metadata)
Call g.CreateEdge(root, g.GetElementByID(tree.right.uuid))
End If
End Sub
End Module最后,将上面的二叉树构建方法和网络图对象导出模块进行组装一下,就可以构建出一个二叉树聚类可视化的工具函数:
''' <summary>
''' make OTU tree graph via cosine correlation method
''' </summary>
''' <param name="otus"></param>
''' <param name="equals"></param>
''' <param name="gt"></param>
''' <param name="env"></param>
''' <returns></returns>
<ExportAPI("makeTreeGraph")>
<RApiReturn(GetType(NetworkGraph))>
Public Function makeTreeGraph(<RRawVectorArgument> otus As Object,
Optional equals As Double = 0.85,
Optional gt As Double = 0.6,
Optional rank_colors As TaxonomyRanks = TaxonomyRanks.Phylum,
Optional env As Environment = Nothing) As Object
Dim pull As pipeline = pipeline.TryCreatePipeline(Of OTUTable)(otus, env)
Dim OtuHashset As New List(Of OTUTable)
If pull.isError Then
Return pull.getError
Else
For Each otu As OTUTable In pull.populates(Of OTUTable)(env)
otu = New OTUTable(otu)
otu.ID = otu.ID.MD5
OtuHashset.Add(otu)
Next
End If
Dim graph As NetworkGraph = OtuHashset.BuildClusterTree(equals, gt)
Dim nodes As Node() = graph.vertex.ToArray
Dim labels As String() = nodes.Select(Function(v) v.data(rank_colors.ToString.ToLower)).ToArray
Dim colorset As New CategoryColorProfile(labels.Distinct, colorSet:="paper")
For i As Integer = 0 To nodes.Length - 1
nodes(i).data.color = New SolidBrush(colorset.GetColor(labels(i)))
Next
Return graph
End Function基于上面所构建的二叉树图的导出代码,我们可以导入makeTreeGraph函数用于R#脚本编程测试:
require(GCModeller);
require(igraph);
imports "OTU_table" from "metagenomics_kit";
# read metabolic annotation embedding data as OTU table
let data = read.OTUtable("metabolic_embedding.csv");
let graph = OTU_table::makeTreeGraph(
data |> OTU_table::impute_missing(),
# use cosine 0.9 cutoff for make clustering
equals = 0.9
);
# export tree network graph to file
igraph::save.network(graph |> compute.network(), "./metabolic_tree/");二叉树聚类可视化测试
运行上面的测试脚本,程序导出了两个表格:network-edges.csv表格存储着我们所构建的二叉树的树枝的信息,也就是节点间的网络边连接信息。而nodes.csv表格存储着基因组的节点信息,包括基因组的唯一编号,物种树信息,以及按照phylum层级进行节点染色的颜色代码结果。现在,我们打开Cytoscape程序,然后通过【File】菜单导入network-edges表格,然后再导入nodes信息表格。之后我们按照相应参数设置好样式映射,最后通过顶部的菜单【Layout】中选择【yFiles Circular Layout】布局,我们就可以将我们的二叉树在cytoscape软件中可视化出来。

我们在Cytoscape程序中进行可视化我们的基因组向量化嵌入结果,可以发现相同颜色的基因组节点几乎都相互据集在一处。这个可视化结果同样支持说明我们对基因组中的代谢酶的注释结果信息进行TF-IDF向量化嵌入的结果可以比较好的将物种基因组进行区分开来,这些向量化嵌入后的结果数据用于下游的机器学习分析可以得到比较好的模型结果。

- 单细胞视角下的微生物基因组代谢酶嵌入分析 - 2026年2月25日
- 基因组代谢酶层级嵌入 - 2026年2月23日
- 基因组功能吉布斯LDA主题建模 - 2026年2月23日



No responses yet