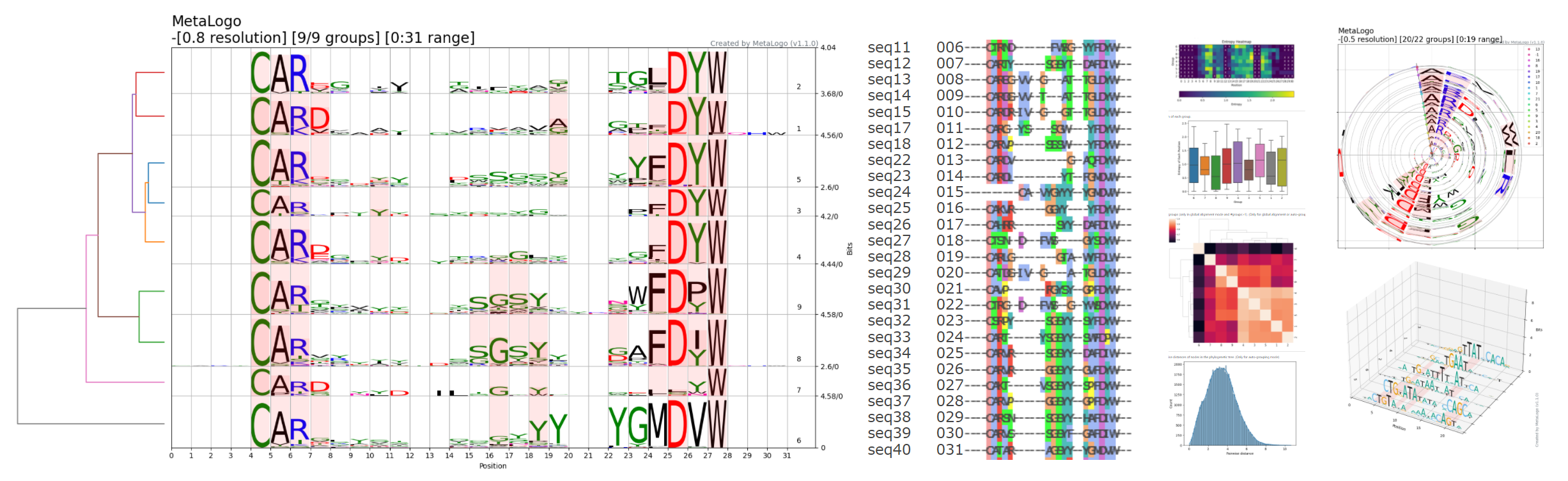
估计阅读时长: 23 分钟Sequence Logo 是一种可视化 DNA 或蛋白质序列保守性的图形表示方法。每个位置(列)上的字母堆叠高度代表该位点的信息含量(以 bits 为单位),而每个字母的高度则与其在该位点出现的频率成正比。高信息量的位置字母堆得高,低信息量的位置则矮甚至接近零。Sequence Logo的绘制遵循信息熵原理,我们可以很直观的通过某一个位置的总高低来了解该处位置的信息含量有多少,高信息量的位置,字母堆的高,一般会出现某一个字符特别高,表明该处非常保守。 位置权重矩阵(Position Weight Matrix, PWM)是描述基因组调控因子结合位点序列模式的核心模型。它通过统计在结合位点序列中每个位置上各核苷酸(或氨基酸)出现的频率,来量化该位置对不同碱基的偏好程度。PWM通常以矩阵形式表示,行对应核苷酸(A、C、G、T/U),列对应序列中的位置,矩阵元素即为该位置该核苷酸相对于背景的权重得分。这一模型简洁且易于计算,因此在转录因子结合位点(TFBS)等调控元件的识别和表征中被广泛采用。 Order by Date Name […]
估计阅读时长: 34 分钟在前面写了一篇文章来介绍我们可以如何通过KEGG的BHR评分来注释直系同源。在KEGG数据库的同源注释算法中,BHR的核心思想是“双向最佳命中”。它比简单的单向BLAST搜索(例如,只看你的基因A在数据库里的最佳匹配是基因B)更为严格和可靠。在基因注释中,这种方法可以有效减少因基因家族扩张、结构域保守等原因导致的假阳性注释,从而更准确地识别直系同源基因,而直系同源基因通常具有相同的功能。在今天重新翻看了下KAAS的帮助文档之后,发现KAAS系统中更新了下面的Assignment score计算公式: We define a score for each ortholog group in order to assign the best […]
估计阅读时长: 5 分钟将复杂的生物学过程拆解为单元化学反应,是进行定量模拟的基石。转录是基因表达调控的关键环节,决定了细胞在特定时间、特定环境下合成哪些蛋白质,对生命活动至关重要。最近的工作中需要将原本非常粗糙的虚拟细胞转录事件模型拆解为更加细分化的多步骤生物化学过程,以适应针对细胞群落生长的建模计算。下面为我将原核生物的转录过程拆解为一系列可以用化学式表示的单元步骤的结果。 在介绍这些分步骤之前,我们会需要首先来定义一下模型中会用到的各种“化学物质”(分子和复合物): RNAP: RNA聚合酶全酶(包含核心酶和σ因子)。 DNA: 基因组DNA双链。 DNA_P: 包含启动子区域的DNA。 DNA_T: 包含终止子区域的DNA。 NTP: 核糖核苷三磷酸(ATP, UTP, GTP, CTP的统称)。 PPi: […]



[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?
谢老师,写快点呀,在看着你更新文章呢。
[…] 最近的工作中我需要按照之前的这篇博客文章《基因组功能注释(EC Number)的向量化嵌入》中所描述的流程,将好几十万个微生物基因组的功能蛋白进行酶编号的比对注释,然后基于注释结果进行向量化嵌入然后进行数据可视化。通过R#脚本对这些微生物基因组的蛋白fasta序列的提取操作,最终得到了一个大约是58GB的蛋白序列。然后将这个比较大型的蛋白序列比对到自己所收集到的ec number注释的蛋白序列参考数据库之上。 […]
[…] […]